在蛋白与DNA的结合研究中,转录因子与靶基因的具体结合位点以及序列是研究者关注的重点之一。基于Homer,我们能够将测序得到的peak的保守序列(motif)进行预测,从而挖掘研究样本中转录因子的结合特征。
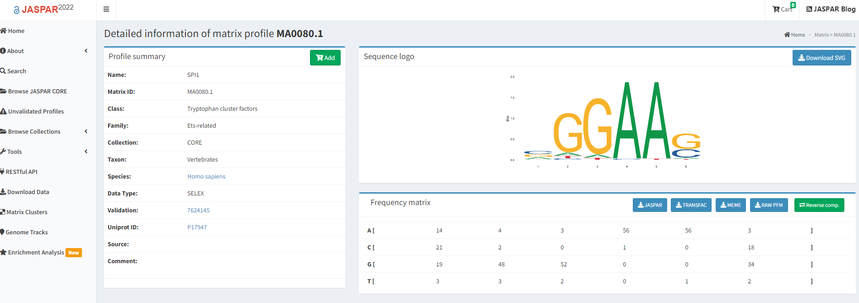
如何将分析得到的motif(图1A)与peak进行对应,找到其在peak上的真实定位和序列,便于进一步深度挖掘和验证,或者直观点,对于某个转录因子的motif(例如来源于Jaspar数据库,图1B),想知道在感兴趣的序列中(比如某个基因的promoter区域)是否存在对应序列;该如何利用非生信手段进行分析呢。小编在这里介绍一个一键式操作的在线软件——MEME_FIMO,帮助各位达到这个目的。

图1B
1、拿到motif的矩阵信息:
这个软件需要输入motif的矩阵。对于前期做了ChIP/CUT&Tag/DAP-seq实验的老师,矩阵可以直接从Homer的分析结果中得到(图2A);对于motif信息来自于预测或数据库,矩阵也可以从数据库中进行下载(图2B)。


图2A
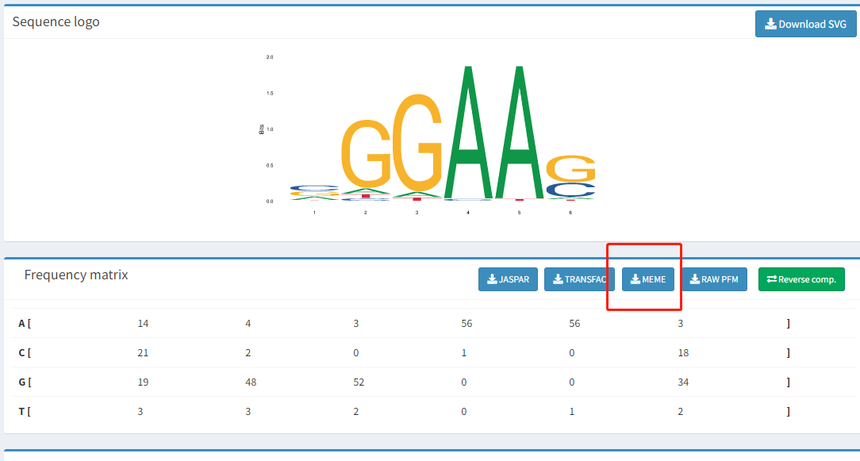
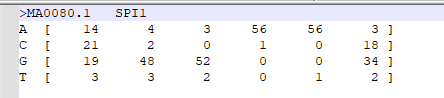
图2B
https://meme-suite.org/meme/tools/fimo
(2)将关注的启动子区序列和motif序列进行上传或者粘贴(需要fasta格式),见图3,点击Start Search箭头:匹配的显著性(可自行调整,默认为p<1E-4)

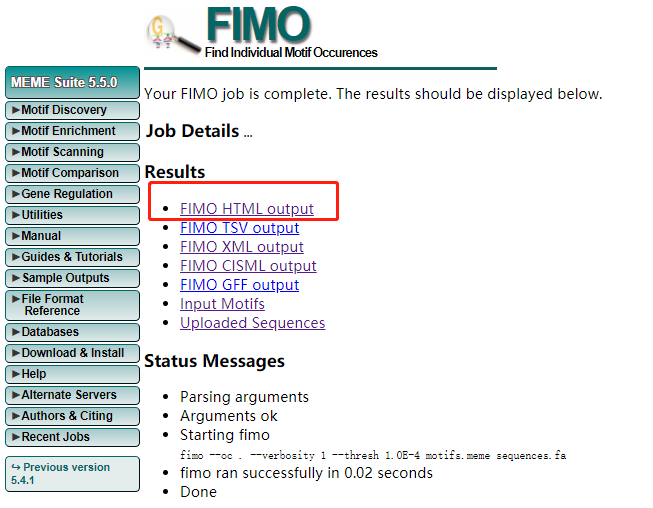
(3)等待输出结果(建议保存收藏栏,这样关闭页面后可以再次从收藏栏里点开找到结果)
想直接在网页上进行查看的话可以点击FIMO-HTML,【SECTION I: HIGH-SCORING MOTIF OCCURENCES】这一部分就是FIMO基于motif与目标序列提供的匹配信息,比较重要的信息为“Start-end”(motif匹配序列位于目标序列的位置)、“p/q-value”(预测的显著性)、“Match Sequence”(目标序列上与motif匹配上的实际序列)。老师们可以根据匹配同源性与显著性两个条件进行筛选。若结果较多,可在输入序列的时候将显著性调整的更严格一点。
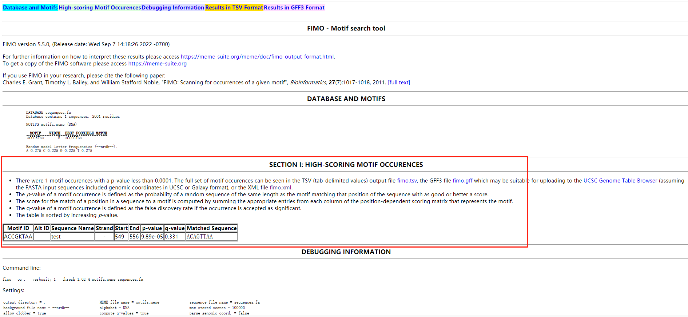
怎么样,是不是很简单?这种“傻瓜式”的操作完全避免了老师需要使用生信手段进行预测的繁琐步骤,贴心地一步到位!当然了,这个软件可不止只有这个小小的功能,还有很多其他方便快捷地motif信息挖掘手段等待着各位老师,感兴趣的老师可以去研究一下哟~