NEWS
新闻资讯
|
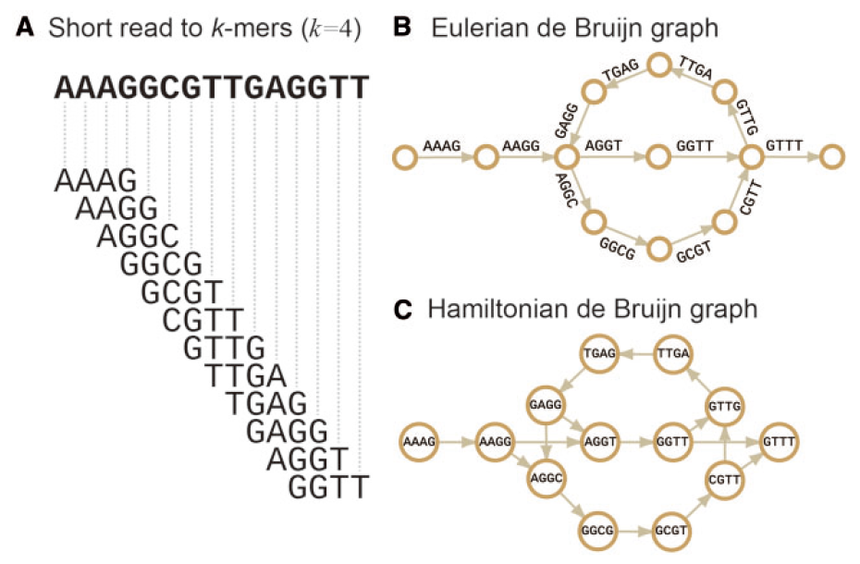
重测序专题(二)| 不断完善的参考基因组对于参考基因组来说,越完善对后续的分析帮助越大。举个例子,当我们分析出基因组某个位置有一段插入序列的时候,通常会认为该位置有一个潜在的结构变异,如果刚好位于一个功能元件上,如增强子或TF结合位点,我们会兴奋的认为久久没有进展的课题终于看到了一丝曙光。但经过PCR扩增并测序验证之后,一盆凉水浇了下来:样间无差异,最后发现是参考基因组错了。于是轻则中午少吃两口饭,重则晚上辗转反侧,对自己整个人生开始反思。可见,正确而完善的参考基因组不仅对于我们的后续研究关系很大,甚至会影响我们的身心健康。 接上一期《重测序专题(一)| 一文了解测序技术的发展》文章,全基因组重测序对单个基因组研究来说,使参考基因组更加完善是重要的研究方向之一。随着测序成本的下降和技术的进步,增加测序深度,更换拼接算法,换用更长读长的测序技术以及多种技术结合等方法,是使参考基因组逾加完善的有效手段。 众所周知,除了病毒和细菌等低等生物外,真核生物的基因组通常都比较大(如人类基因组约有30亿个碱基对),而由于技术限制,别说整条基因组了,我们目前能够测序的最长片段也仅有150kbp左右,于是鸟枪法应运而生。最早利用鸟枪法测序的完整基因组为病毒[1],目前绝大部分的基因组均是利用鸟枪法测序的。 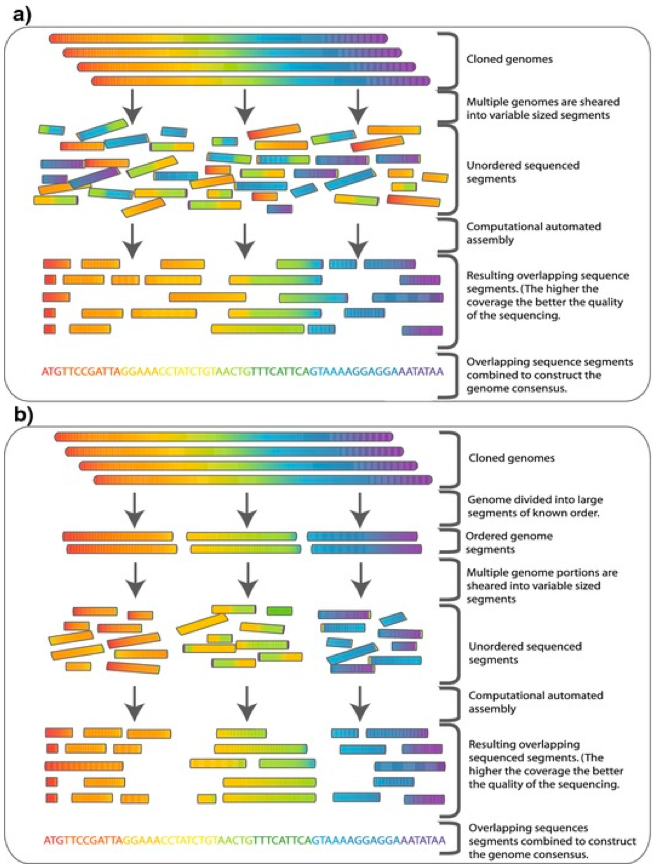 图1. 鸟枪法基本原理 该方法的基本原理如图1所示,原本很长的基因组大片段被切割成一个个小片段,然后根据不同小片段的重叠部分重新拼接成完整的大片段。为了能够重新拼接成基因组,足够丰富的小片段,也就是足够的测序深度是必须的。 那什么是测序深度?简单来说就是基因组的每个碱基被测了几次,例如50×的测序深度就是指基因组的每个碱基平均被测了50次。由于成本和技术的限制,最早的人类基因组草图平均测序深度仅有5.11×[2],而最新的人类T2T-CHM13基因组,仅PicBio HiFi单独一种测序策略就用了30×[3]。 对于重测序来说,后续的不同分析,所需的测序深度也不相同。以单核苷酸变异(single-nucleotide variants, SNV)检测为例,通常30×是普遍认可的测序深度标准,但也有文章认为50×才能保证在覆盖95%基因组的情况下得到可靠的SNV和小的indel[4]。 有了足够的测序深度数据,接下来的挑战就是如何将这些杂乱无章的测序小片段拼接成完整基因组。当有参考基因组的情况下,我们只需要将这些小片段根据序列相似性比对上去即可,这大大减少了计算难度。但如果参考基因组本身质量不高,从头组装(de novo)一套高质量参考基因组就势在必行,而合适又高效的算法对de novo组装尤其重要。接下来我就给大家介绍一下常见的de novo基因组算法及相应软件。 De Bruijn graph德布鲁因图(de Bruijn graph),是一个有方向的,用来展示符号之间重叠关系的示意图。当我们拿到一条序列的时候,首先要将这条序列分成长度为k的小片段,称之为k-mer,相邻片段会重叠k-1个碱基,总共将得到L-k+1个k-mer (L为序列长度)。图2A所展示的就是当k=4的时候,k-mer是如何分割的。因此,我们就将基因组拼接问题转换成寻找最优路径的问题。根据对顶点(nodes)和边(edges)认识的不同,de Bruijn graph被分为了Hamiltonian de Bruijn graphs 和 Eulerian de Bruijn graphs两种类型,如图2B和图2C所示。利用de Bruijn graph算法进行de novo组装的常用软件有很多,如ALLPATHS-LG[5]、SOAPdenovo[6]以及SparseAssembler[7]等。
图2. De Bruijn graph示意图和两种不同的分类 ALLPATHS-LG基于Eulerian de Bruijn graph,被认为是短reads最准确的拼接软件。与其他基于Hamiltonian de Bruijn graph的软件不同,ALLPATHS-LG在最后一步之前都不会删除错误或冗余路径,因为它们有可能是正确路径。这需要相对较大的内存,对于大型脊椎动物基因组的拼接可能需要数周时间。尽管ALLPATHS-LG最初仅设计用于Illumina短reads,但也可以用于小型细菌基因组组装的PacBio长reads。SOAPdenovo2实现了一种基于稀疏 k-mer 的算法,并将组装人类全基因组所需内存减少到35 GB,并且其装配质量可与 ALLPATHS-LG相媲美。该软件的强项在于,从头组装的每个步骤(例如纠错、contigs或scaffolds组装、gap填充)都可以根据情况需要单独执行。SparseAssembler的一大优势是将所需内存减少了一个数量级。对于人类14号染色体的组装,SparseAssembler在k = 54的条件下,仅使用了3GB的内存。而其他使用普通k-mer策略的软件需要高达30GB-50GB内存(如Velvet为37 GB,ABySS为49 GB以及SOAPdenovo为30GB)。 String graphString graph在维基百科中是这么描述的 :“In graph theory, a string graph is an intersection graph of curves in the plane; each curve is called a "string”.” 翻译过来就是: 在图论中,弦图是在平面上由曲线连接起来的交叉图,而每条曲线被称为“弦”。最早提出string graph并运用在基因组组装的是2005年来自美国加州大学伯克利分校的E. W. Myers[8],图3展示的就是string graph的基本原理图。简单来说,它利用所有reads的overlap区域计算拼接成最终基因组。利用string graph算法的常用软件有:SGA[9]等。
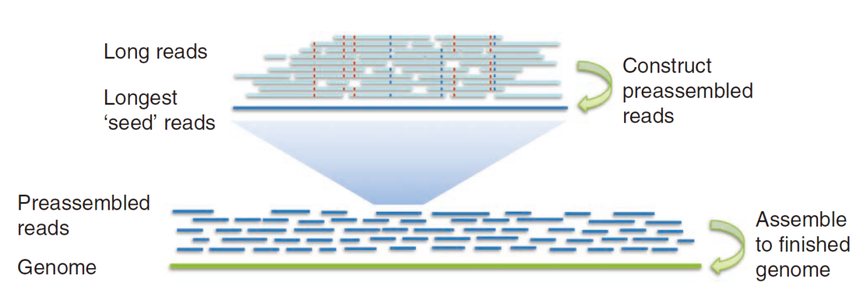
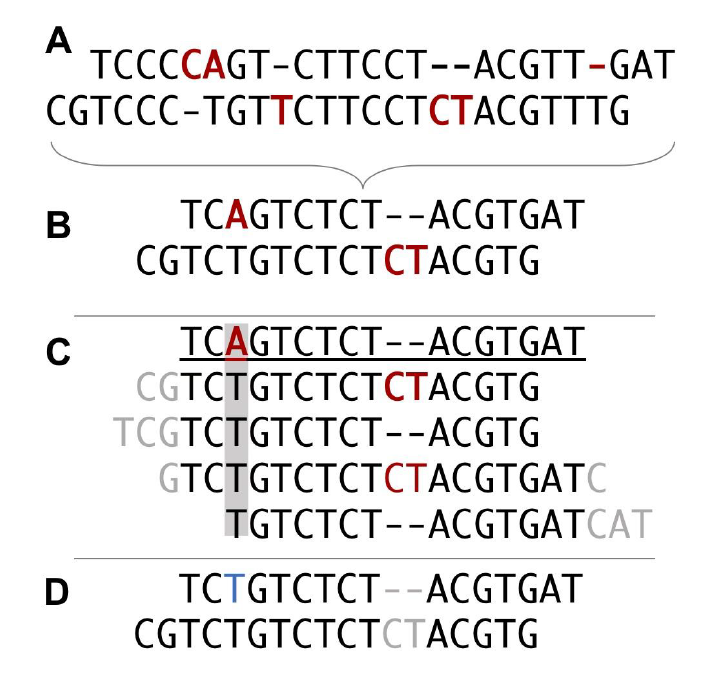
图3. String Graph的基本原理 SGA (String Graph Assembler)是基于overlap的string graph模型的装配软件,由于低内存和支持并行计算,并且不需要进程间通信,SGA 是第一个可以在低端计算集群上进行大基因组(如哺乳动物)组装的软件。 与de Bruijn graph 方法相比String graph不需要将reads切割成较短的k-mer,当长的reads被切割成很短的k-mer的时候,会丢失掉原本的位置关系信息。当reads仅有上百bp的时候,丢失的位置信息我们尚可以接受,但当一条10kb的reads被切割成仅有几bp或几十bp的k-mer时,丢失的信息就是我们无法接受的。因此,第三代测序技术产生的长reads已经不适用现有的基于de Bruijn graph的拼接算法,我们更多使用的是基于reads间overlap的算法,string graph就是其中之一。 Overlap–Layout–Consensus (OLC)OLC算法在NGS的短序列中毫无用武之地,而在有较长reads的三代测序序列中却如鱼得水。目前的三代测序平台主要以PacBio和ONT为主,而每个平台都有对应的分析软件。例如基于PacBio的SMRT平台的 HGAP[10], FALCON[11], HiCanu[12]等,基于ONT平台的ProeSeq[13], Shasta[14]等。 HGAP的装配原则被称为分级基因组组装(hierarchical genome assembly process),如图4,它是以SMRT平台得到的最长reads为种子,然后将剩余reads比对到该种子reads上,如此一级一级的组装,最终组装为完整基因组。FALCON同样是基于分级基因组组装原则,但其优化了相关算法,可以应用于杂合基因组组装。而HiCanu是基于Canu[15]的修改版,主要应用于PacBio的HiFi平台产生的reads进行基因组组装,该平台产 生的数据以长reads(>10kbps)和高单碱基正确率(>99.9%)为主要优势。但HiFi也不是完美的,它会在均聚物序列或简单重复序列区域出现小的插入缺失。因此,如图5 所示HiCanu的解决方案是首先将重复区域压缩成单个碱基(例如,“AA…”变为“A”),然后和其他序列进行比对从而矫正测序错误,得到高度一致序列后再进行组装。
图4. HGAP的装配原则
图5. HiCanu的压缩矫正原理 PoreSeq是一个基于Python语言开发的开源软件,主要利用ONT的MinION平台所生成的数据进行基因组的组装、变异分析等。Shasta的主要特点是大大缩短了基因组组装时间,在9天时间内共组装了11个高度连续的人类基因组。另外,还有一些软件可以利用两个不同平台的数据,如Canu[15]、 wtdbg2[16]、HINGE[17]等。 有的时候我们发现,仅仅利用三代测序技术产生的长reads在经济和技术上并不足以装配出高质量的基因组。因此我们需要一些其他技术来辅助基因组的装配,如利用二代测序短reads进行测序质量矫正,利用Hi-C或基于Bionano的光学图谱进行基因组的辅助拼接,以及利用ONT的超长reads (>100kb) 技术进行gap的填补等。(由于篇幅有限,我们会在之后的文章中专门介绍这些技术原理以及应用)。 随着拥有高质量高连续性的T2T基因组的物种增多,这为我们的科研工作提供了很大的便利,也为后续一系列的分析工作打下了坚实的基础。在下一篇文章中我们将会介绍全基因组重测序在群体中的应用,敬请期待。 |