NEWS
新闻资讯
|
文献解读 ▏ATAC-seq和RNA-seq揭示了苹果应对干旱的染色质可及性和基因表达的动态变化
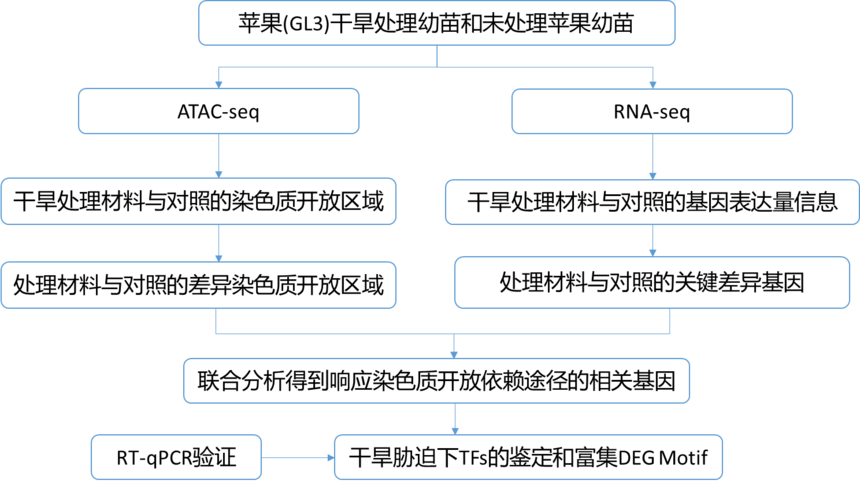
题目:Integrating ATAC-seq and RNA-seq Reveals the Dynamics of Chromatin Accessibility and Gene Expression in Apple Response to Drought 期刊:International Journal of Molecular Sciences 材料:苹果(GL3)干旱处理幼苗、苹果幼苗 植物的抗旱性受到多种信号通路的影响,这些信号通路涉及多种转录因子、多种靶基因和多种类型的表观遗传修饰。对干旱的表观遗传修饰的研究主要集中在DNA甲基化和组蛋白修饰,而对染色质重塑的研究较少。染色质可及性的变化可以通过影响RNA聚合酶结合和各种调节因子在植物的非生物胁迫中发挥重要作用。然而,苹果在干旱期间染色质可及性的变化尚不清楚。作者通过ATAC-seq和RNA-seq分析了干旱条件下与苹果(GL3)基因表达相关的染色质可及性区域。干旱处理和对照之间的差异分析确定了23466个染色质可及性上调peak和2447个下调peak。干旱诱导的染色质可及性改变基因主要在代谢、刺激和结合途径中富集。RNA-seq和ATAC-seq联合分析鉴定了240个在干旱条件下染色质可及性较高且基因表达增加的基因,这些基因可能在干旱响应过程中发挥重要作用。其中,共鉴定了9个转录因子基因,包括ATHB7、HAT5和WRKY26。这些转录因子基因在不同染色质可及性基序结合位点上差异表达,这些位点可能通过调节下游基因参与苹果对干旱的反应。该研究为苹果在干旱胁迫下染色质的可及性提供了参考,其结果将有助于后续对染色质重塑物和转录因子的研究。 02 技术路线
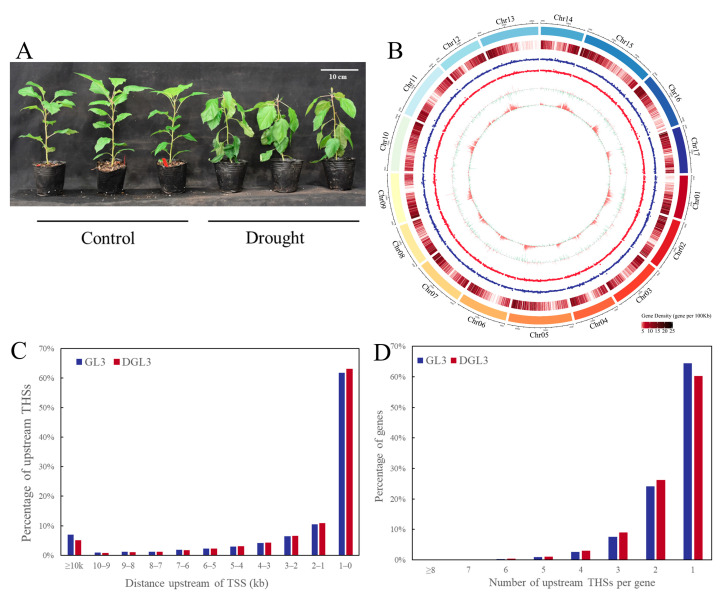
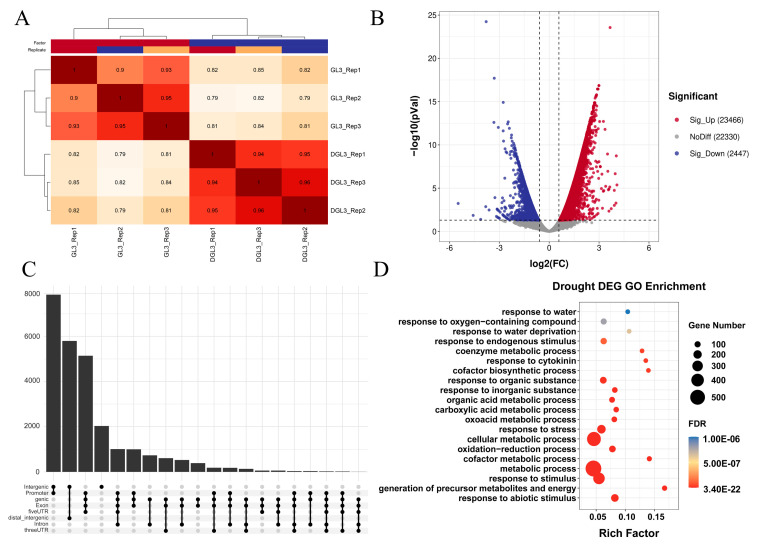
03 研究成果 作者将苹果(GL3)幼苗组织培养植株进行12天的干旱处理(DGL3);处理后的叶片明显表现出缺水下垂表型(图1A)。然后作者使用DGL3和GL3叶片进行ATAC-seq和RNA-seq(图1B)。结果显示,干旱组和对照组开放染色质的分布相似,只有几个不同的区域。差异开放染色质峰在基因密度较高的区域明显更高,干旱处理后显著更多的差异峰上调而非下调。差异表达基因的分布相对于开放染色质区域均匀分布,上调和下调基因的数量没有显著差异(图1B)。
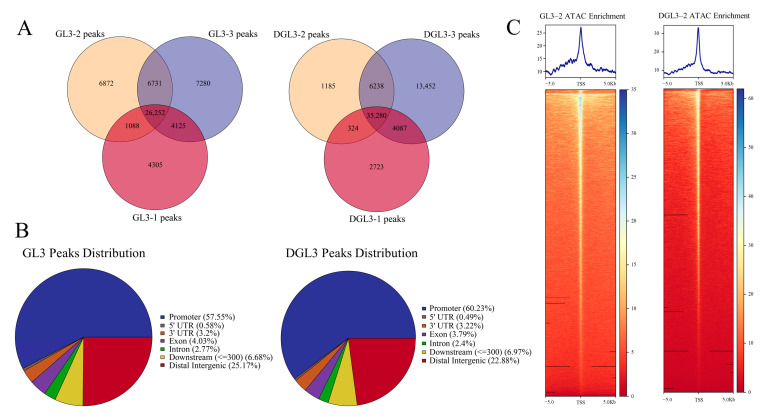
为了获得更高置信度的信号,作者选择了三个生物学重复的交集作为THS的高置信度信号进行进一步研究(图2A)。与拟南芥和水稻中发现的相似,苹果中超过50%的THS分布在启动子上游3.5kb的区域(启动子定义为TSS-3500至+100bp区域)。约25%的THS位于基因间区域,6%位于下游区域,6%在基因体区域(外显子和内含子)。干旱后,启动子区域的THS略有增加,但与对照相比没有显著差异(图2B)。 GO分析对对照和干旱处理中与THS相关的基因进行功能富集。结果表明,在两种处理中,与代谢、发育和应激刺激相关的基因富集,这表明大多数可获得的染色质对应于为正常植物生长而主动转录的持家基因。TSS附近5kb的THS热图和信号图分析结果表明,在基因的TSS附近有许多THS,表明在对照和干旱处理中,TSS附近的染色质可接近性更强(图2C)。两种处理中开放染色质的相似分布表明,开放染色质区域是正常植物生长和发育所必需的途径。
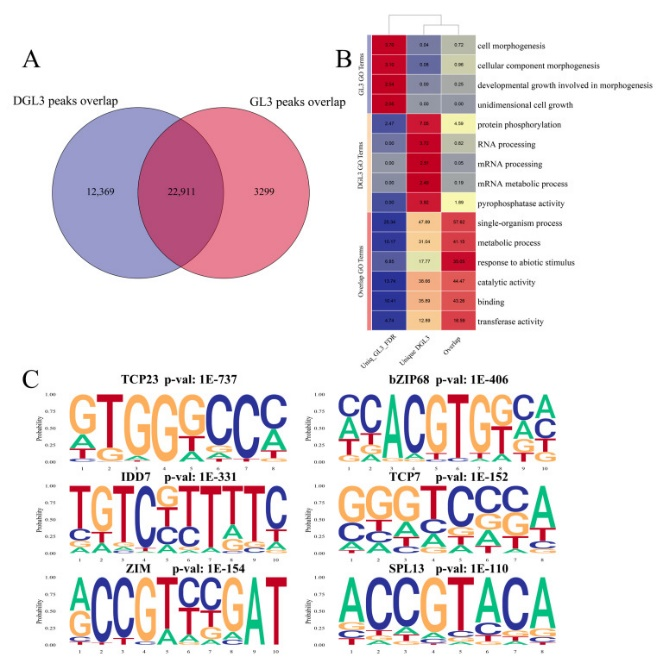
图2.GL3和DGL3干旱处理后ATAC信号的表征 2、对照和干旱处理中ATAC峰的motif分析和功能分析 对照和干旱处理的THS的交集分析发现22911个THS,这些THS可能表明在正常生长条件下转录的基因和植物发育所需的基因(图3A)。干旱和对照处理分布具有特异性12369和3299个THS。功能分析表明,与对照特异性THS相关的基因主要富集在生长和发育相关的途径中。与干旱特异性THS相关的基因大多富含RNA途径和蛋白质磷酸化(图3B)。富集结果表明,干旱条件下染色质的开放可以影响RNA过程和蛋白质磷酸化。 作者进一步寻找与干旱相关途径相关的基因。分析显示,12369个THS与10457个基因相关。这10457个基因包括710个转录因子基因,包含65个AP2/EREBP家族蛋白、91个MYB家族蛋白等和其他干旱相关转录因子。由于蛋白质磷酸化是干旱处理下信号转导的常见和关键途径之一,作者对富集结果中309个与蛋白质磷酸化相关的基因进行了进一步分析。在这些基因中,有11个是Ca2+依赖性蛋白激酶(CPKs/CDPKs),它们识别Ca2+信号以磷酸化保卫细胞中的ABA效应物。ABA可以诱导H2O2,其抑制ABA-INSENSTIVE1(ABI1)和ABI2,从而减轻ABI2对保卫细胞过氧化氢抗性1(GHR1)的抑制。ABI2也被发现为蛋白质磷酸化相关基因。309个与蛋白质相关的基因与蛋白磷酸化相关的9个基因包括7种钙调神经素B样蛋白相互作用蛋白激酶(CIPKs)、5种丝裂原活化蛋白激酶激酶(MAPKKKs)、3种盐过度敏感蛋白3(SOS3)相互作用蛋白和在信号转导中具有重要作用的丝裂原激活蛋白激酶3(MPK3)。结果表明,干旱诱导的特异性染色质开放可能在信号转导中发挥重要作用。 重叠THS的基序的富集分析确定了其他转录因子基因,如TCP23、TCP7、bZIP68、IDD7、ZIM和SPL13(图3C)。先前的研究报道,TCP23和TCP7可能参与开花时间控制和植物发育。IDD7在植物的生长和发育中起着负调节作用。SPL13、bZIP68和ZIM参与不同植物的胁迫响应和发育。这些重叠的THS相关转录因子在参与生长和发育的途径中明显富集,并可能在干旱处理过程中发挥额外作用。
图3.干旱处理期间ATAC-seq的功能富集 3、差异分析揭示干旱处理后染色质可及性和转录体的动态变化 差异分析结果表明,干旱后DAR的数量(23466个峰值)显著大于下调的DAR数量(2447个峰值)(图4B),这一发现与先前在水稻中的研究结果相似。DAR的基因组分布与单一处理条件下THS的分布相似(图2B),大多数信号位于启动子和基因间区域(图4C)。最后,作者检查了DAR附近基因的功能富集,发现上调和下调基因在GO方面都显著富集,涉及代谢、刺激和结合。 转录组热图和差异火山图分析表明,下调基因的数量(658个)略多于上调基因的数量,三个生物重复序列明显聚为一类。DEG的总数(1243)显著低于DAR的变化数量(图4B)。DAR主要是上调的基因(图4B)。然而,下调的DEG数量略有下降在转录组数据中,DAR的变化比DEG更剧烈。对于GO富集分析,DEG在干旱相关GO术语中显著富集,包括非生物胁迫、代谢过程和水响应(图4D),表明DEG与干旱高度相关。 干旱条件下的转录组差异分析表明DEG与DAR显著不同。这可能表明,作为主要反应的染色质可及性的变化可能通过各种调节途径产生不同的影响。RNA-seq和ATAC-seq的联合分析可以筛选对基因表达有显著影响的开放染色质区域。
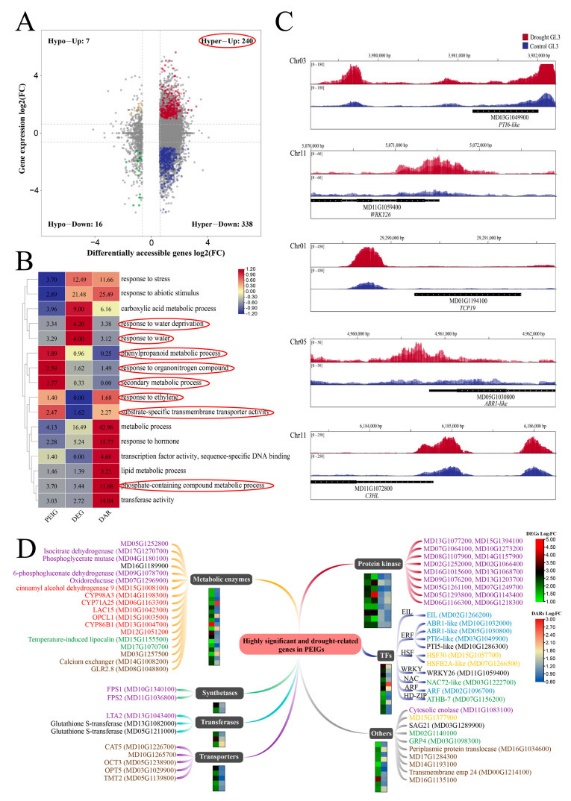
图4.差异ATAC分析结果 4.联合分析揭示了染色质开放依赖途径和相关基因 启动子附近染色质的开放可能通过促进RNA聚合酶II的结合,或通过增强转录激活物或阻遏物的结合来促进基因表达;作者通过ATAC-seq和RNA-seq联合差异分析,鉴定了与表达改变相关的染色质可及性发生干旱诱导变化的基因(图5A)。其中,共有240个基因被鉴定为染色质可及性上调和表达水平上调(图5A),表明这些基因的产物可能在干旱胁迫中发挥作用。根据注释,按基因类型对具有更大启动子可及性和表达增加(PEIG)的基因进行分类。这些PEIG也用DEG和DAR进行功能富集分析(图5B)。DAR在代谢、激素、转移酶活性和DNA结合方面更加丰富,而DEG在水分胁迫等途径中显著丰富。如图5B所示,PEIG在与苯丙类代谢、二次代谢、乙烯反应和有机氮化合物反应相关的途径中更显著地富集。因此,与DAR和DEG相比,PEIG可能在二次代谢和乙烯反应中发挥更重要的作用。作者选择PTI6-like、WRKY26、TCP19、ABR1-like和C3HL作为代表性PEIG,并分析了THS在其启动子区的分布(图5C)。WRKY26和TCP19与非生物胁迫相关,PTI6和ABR1可能参与生物和非生物胁迫。 这些基因可能因染色质可及性的变化而差异表达,这表明它们在苹果的干旱反应中起着重要作用。为了进一步分析PEIG,作者选择了显著富集和水分胁迫相关的PEIG基因进行更详细的注释(图5D)。由于干旱特异性THS相关基因包含许多磷酸化相关激酶,作者还提供了与含磷酸盐化合物代谢过程相关的基因的详细注释。大多数参与代谢酶的基因来源于磷代谢和次级代谢过程,包括苯丙类代谢。这些代谢相关酶含有氧化还原酶和细胞色素相关酶,这些代谢酶可能受干旱条件下苹果染色质可及性的调节。在选定的基因中,包括18种蛋白激酶,这可能表明干旱相关的磷酸化途径可能受染色质可及性的影响。 在转录因子基因中,ERF是乙烯反应因子,在应激反应中起作用。缺水相关的转录因子基因包括NAC72样、ATHB7和代谢酶,这些基因可能参与干旱的反应。 有机氮化合物相关基因和HSF的富集,这些基因先前被鉴定为在非生物胁迫响应中起作用[40]。PEIG中缺水基因的注释结果表明,受开放染色质影响的基因可能参与代谢过程、转录因子调节和蛋白质磷酸化。
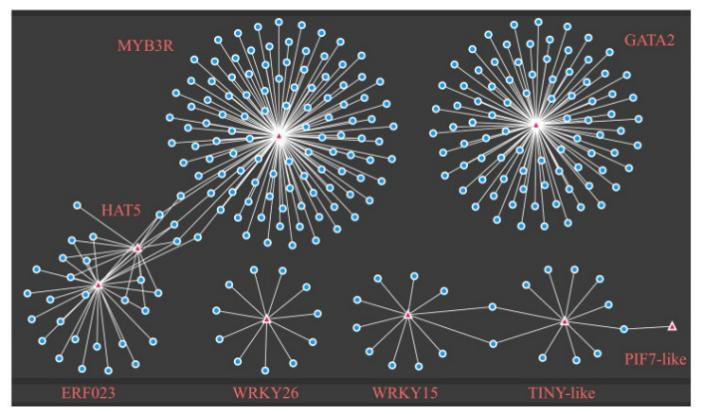
图5. ATAC-seq与RNA-seq的关联分析 作者还重叠了12369个与干旱和DEG相关的THS基因,以鉴定具有干旱特异性表达变化的基因。结果表明,共有326个差异基因与干旱特异性开放染色质相关,热图显示了相似数量的上调和下调基因。对326个干旱特异性染色质可及性相关基因的进一步注释显示,光合作用、电子运输链、茉莉酸代谢过程和木质素代谢过程途径显著富集。 这些基因包括LOX2、LOX3、AOC4、ST2A和PKT3,它们与茉莉酸代谢有关。CAD9、CYP98A3、LAC15、OMT1和过氧化物酶超家族蛋白与木质素代谢有关。茉莉酸和木质素在非生物胁迫过程中具有重要作用。干旱条件下这些基因的特定染色质开放可能调节其差异表达和干旱反应中的潜在功能。 5.识别干旱胁迫下的TFs和富集DEG Motif分析 染色质开放调节RNA聚合酶II和转录因子的结合。通过使用DAR的基序来寻找在干旱处理下具有活性的潜在转录因子,从而鉴定DEG中转录因子的结合位点。 DEG中鉴定出差异表达的转录因子,干旱后分别有23个和16个转录因子表达上调和下调(图6A)。结合DAR的基序,最终获得了9种在干旱条件下具有显著染色质结合基序的差异表达转录因子(图6B)。ATHB7、MYB3R1和ERF023先前显示对干旱有反应。WRKY转录因子家族在植物对胁迫的反应中起作用。PIF7和TINY在干旱胁迫相关途径中被鉴定。具有不同染色质可及性基序的DEG可能在干旱调节途径中起作用。  图6. 差异表达转录因子和差异ATAC信号基序分析 为了确定具有不同染色质可及性基序的差异转录因子的可能调节功能,作者对差异表达基因进行了共表达网络分析。共表达网络分析通过筛选进行,p值<0.01,PCC>0.9(图7)。分析表明,除了ATHB7之外,所有转录因子基因都与差异表达基因相关。详细的功能分析表明,TINY样和WRKY15相关基因在非生物胁迫、外部刺激和氧化胁迫的功能方面更为丰富。GATA2、HAT5和MYB3R1相关基因在光合、能量和代谢途径中明显丰富。与ERF023相关的基因在参与脱落酸、分解代谢的途径中富集,与WRKY26相关的基因与乙烯反应和免疫反应相关。这些因素可能调节这些基因,作为苹果干旱反应的一部分。
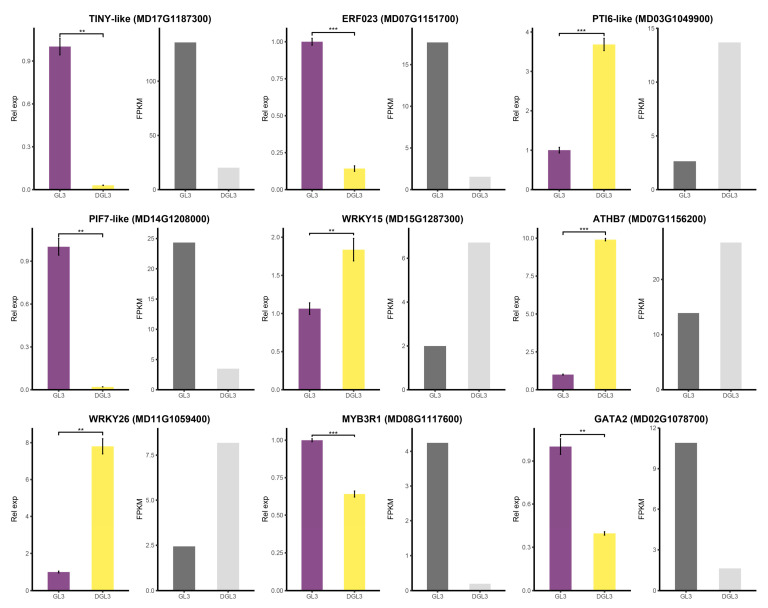
图7.具有不同染色质可及性基序的差异表达转录因子网络的可视化 6.验证干旱处理下响应于染色质可及性改变的基因表达的变化 染色质可及性的改变可能影响基因表达,从而影响植物的抗旱性。为了验证上述结果的可靠性,作者对一些具有染色质可及性变化的差异转录因子进行了RT-qPCR验证。RT-qPCR结果与转录组数据的趋势一致,进一步证明了这些转录因子对干旱的反应(图8)。
图6. RNA-seq和RT-qPCR检测差异表达转录因子的相对表达 04 总结 |