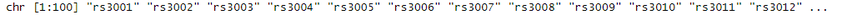NEWS
新闻资讯
|
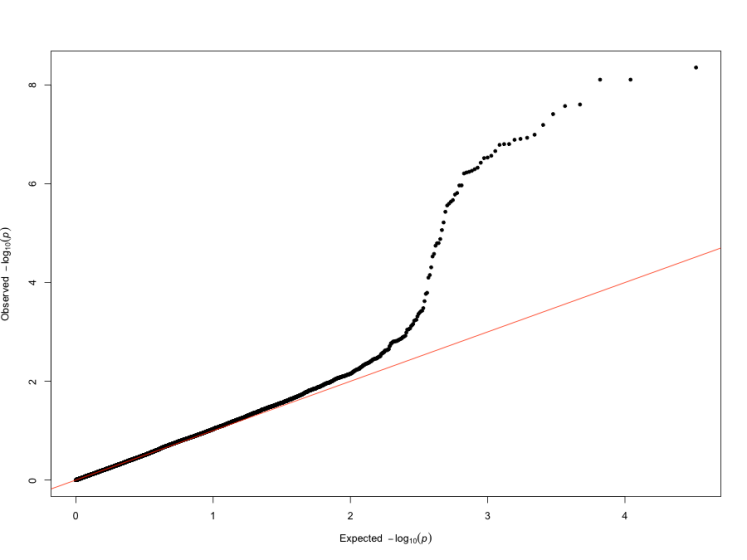
科研绘图|一个R包轻松搞定Manhattan图和QQ图NO.1 简 介 Mahattan图和QQ图都是在遗传学中常用的图形。Manhattan图通常用于展示基因组关联研究(GWAS)的结果,而QQ图则是用来验证GWAS结果是否符合期望分布的工具。 在本文中,我们将使用R语言中的qqman包来绘制这两种图。
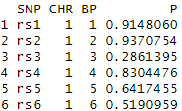
QQ图 NO.2 安装qqman包 首先,我们需要安装qqman包。在R环境中,你可以使用以下代码来安装:install.packages("qqman") 安装完成后,使用以下代码加载包: library(qqman) NO.3 数据准备 在开始绘图之前,我们需要先准备数据。这里我们假设你已经有了GWAS研究的结果,该结果应包含以下信息:SNP名称,染色体位置,基因位置,以及P值。这些数据应该在一个数据框(dataframe)中,每一列的名称分别为SNP、CHR、BP和P。之后的结果,我们使用包中子代的数据进行绘制。 head(gwasResults)
NO.4 绘制Manhattan图 有了数据,我们就可以开始绘制Manhattan图了。在qqman包中,我们使用manhattan函数来绘制Manhattan图。 manhattan(mydata) 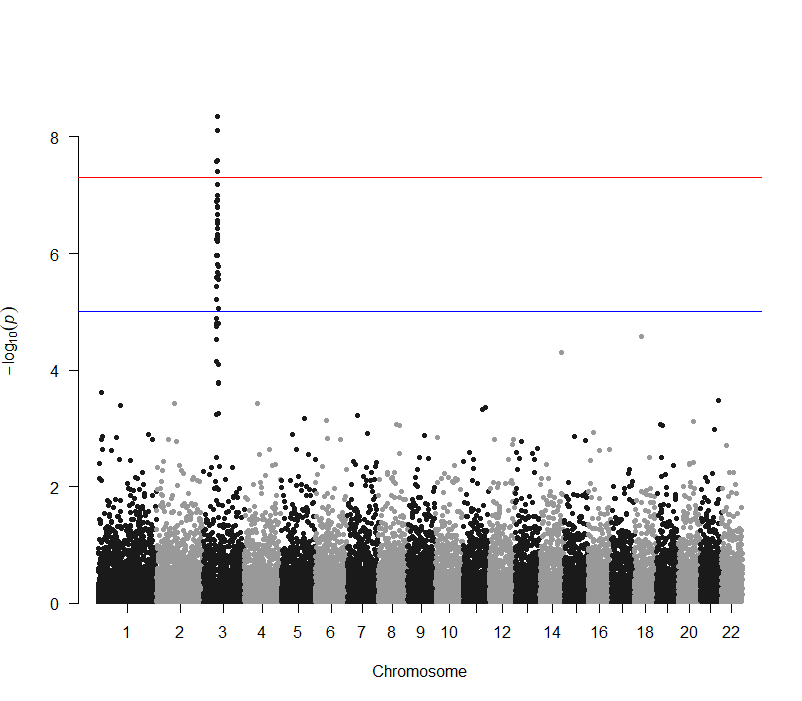 如果想要突出显示某些SNP,可以使用highlight参数。这里我们突出显示最高点附近的100个SNP位点,这些位点存储在snpsOfInterest函数中。 str(snpsOfInterest)
manhattan(gwasResults,highlight = snpsOfInterest) 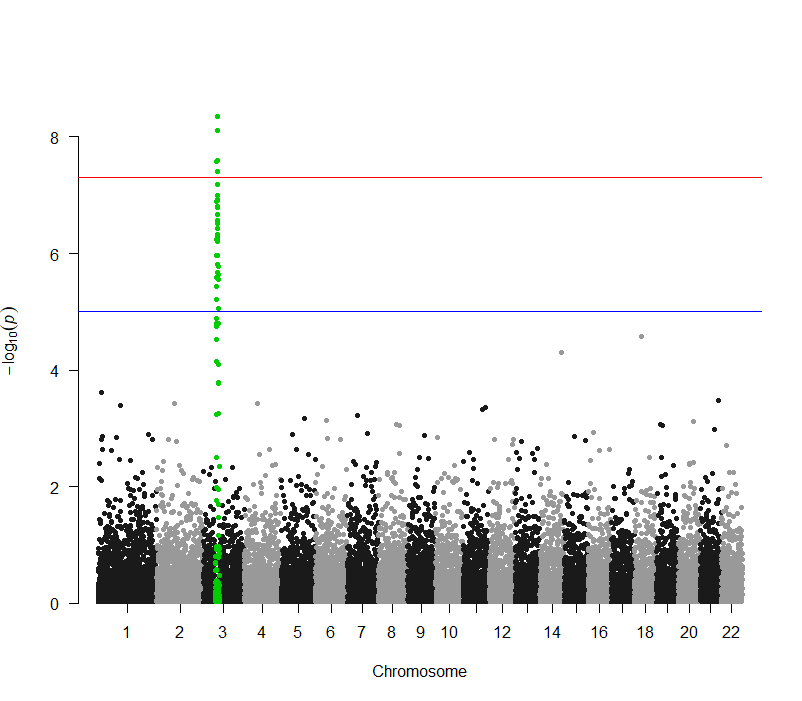 NO.5 绘制QQ图 QQ图的绘制相对简单一些,我们只需要用到GWAS结果中的P值。在qqman包中,我们使用qq函数来绘制QQ图。 qq(gwasResults$P) 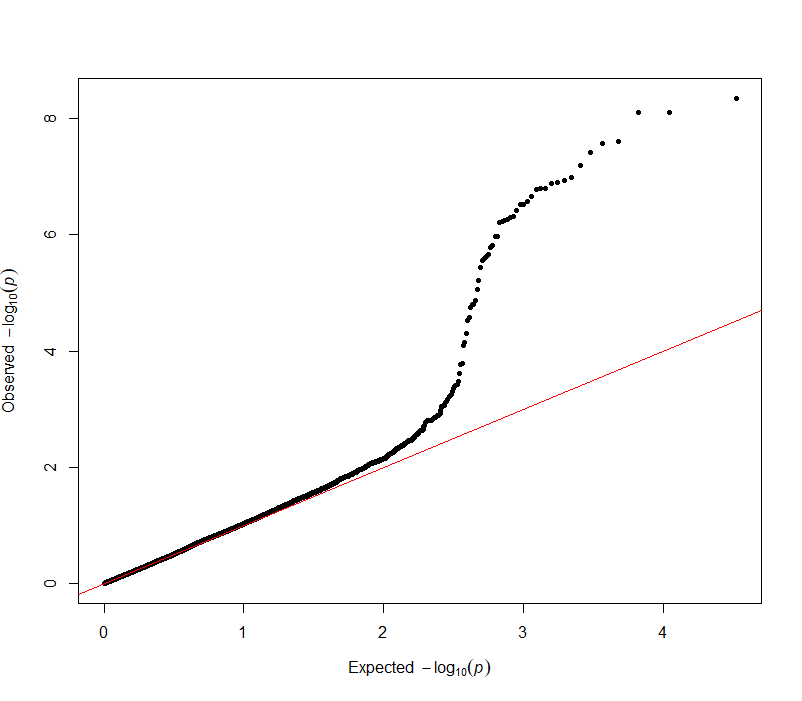 你可以使用main参数来添加标题。 qq(gwasResults$P, main = "QQ Plot of GWAS P-Values") 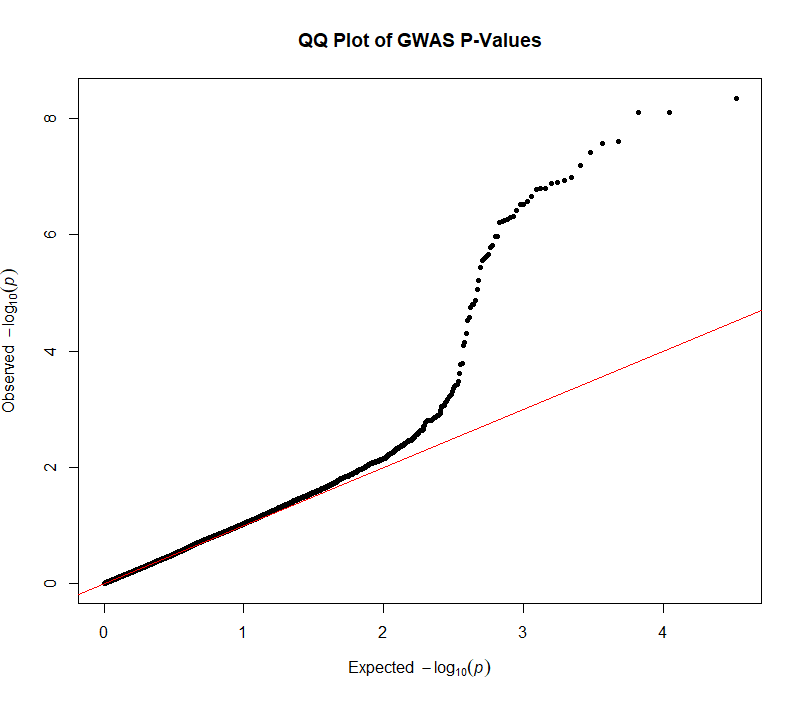 结 论 以上就是在R语言中使用qqman包绘制Manhattan图和QQ图的方法。虽然这个包提供的功能相对简单,但它已经足够用于大部分的GWAS结果可视化需求。如果你需要更复杂的功能,可能需要考虑使用如ggplot2这样的更强大的绘图包。 希望这篇教程对你有所帮助,如果你有任何问题或者需要关于R语言的更多教程,欢迎随时和我们沟通。 了 解 更 多 { 往 期 精 彩 回 顾 } |