NEWS
新闻资讯
|
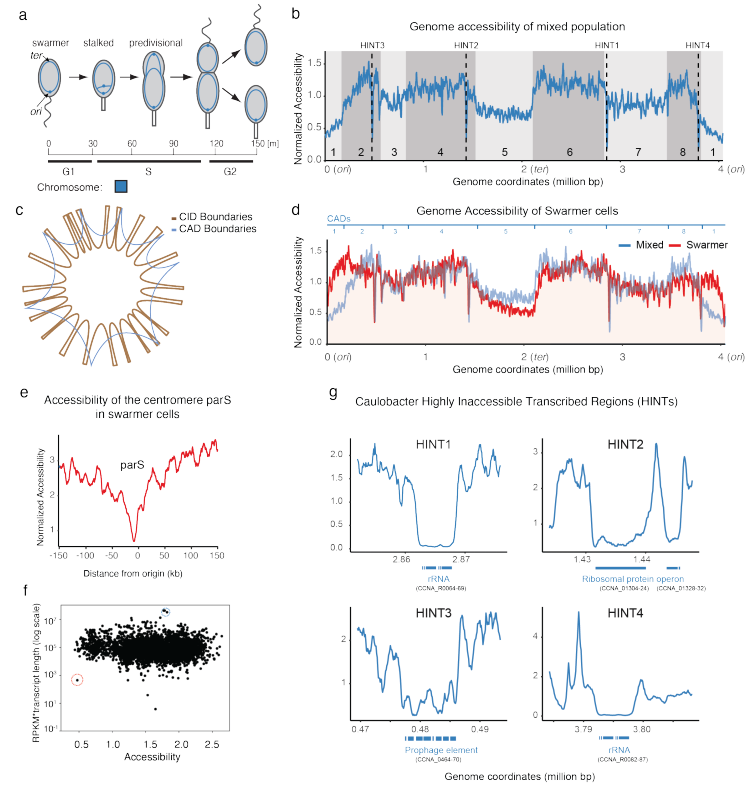
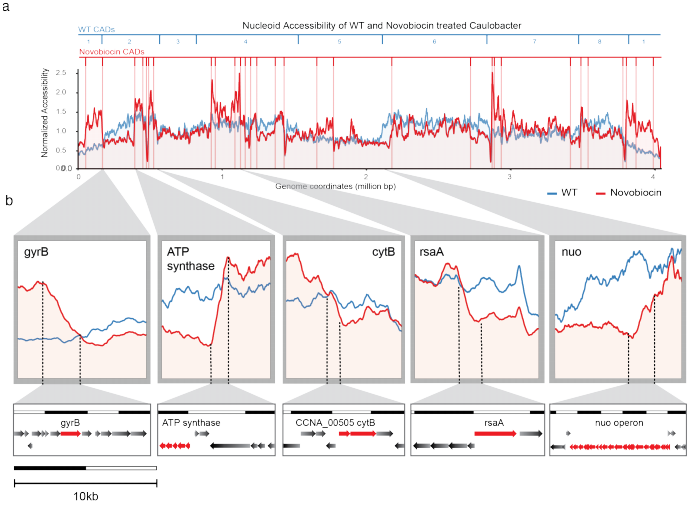
打破旧思维,细菌ATAC-seq展示细菌基因组开放区域特性 在我们常规的认知里,ATAC-seq实验首先需要提取样本完整的细胞核,之后再用Tn5转座酶对核小体区域进行切割。细菌没有完整的细胞核,只有被称为拟核的区域,常规的提核步骤对细菌也不适用。另外,细菌的基因组较小,很难将细菌的基因表达调控与染色质开放区域结合。但是今天我们要分享的文章里,作者开辟性思维将细菌与ATAC结合进行新视角解说细菌的基因调控。 作者有很多开创性思维点,首先是实验部分,因为细菌没有核小体包裹组成的细胞核,所以作者将甲醛交联应用在了本实验中。为什么有此步骤应用,作者给出了理由:一些细菌中已经报道了细菌中存在一些约100 kb的高度自我互作区被称为chromosomal interaction domains (CIDs)。通过甲醛交联,这些互作区形成蛋白核酸结合区域,不会被Tn5酶进行切割。在分析阶段,细菌的ATAC测序结果不能也得不到常规真核生物的测序结果(如核小体分布pattern,较好的TSS分布图,motif预测等等)。文章里作者将染色体开放区域Chromosomal Accessibility Domains (CADs)与细菌染色体互作区域chromosomal interaction domains (CIDs)结合分析,对比这些CADs与CIDs区域,发现了它们的关联点。通过Bac-ATAC-seq实验,作者鉴定出了柄杆菌中有4个Highly Transposase-Inaccessible Transcribed(HINT)regions,对这些区域进行分析,得出了这些HINT区域的基因组可接近区域图谱。当然,作者也对大肠杆菌进行了同样的Bac-ATAC-seq测序及分析,对比了柄杆菌和大肠杆菌开放区域及组成,发现了它们的相同与差异,如柄杆菌和大肠杆菌中基因组非开放区域与转录活性的相关性不高(R2=0.070),还有非开放区域主要是一些核糖核蛋白。 想要了解更多细节,大家可以移步翻阅文章全文: 1. ATAC-seq reveals megabase-scale domains of a bacterial nucleoid(https://www.biorxiv.org/content/10.1101/2021.01.09.426053v1) 2. Dynamic landscape of protein occupancy across the Escherichia coli chromosome(https://www.biorxiv.org/content/10.1101/2020.01.29.924811v3) |