NEWS
新闻资讯
|
文献解读 | 玉米中的DNA和染色质特征测序绘制全基因组增强子图谱
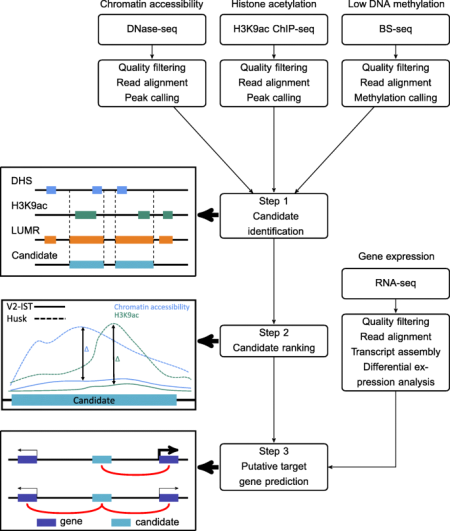
Title: Genome-wide mapping of transcriptional enhancer candidates using DNA and chromatin features in maize Date: 2017年7月 Journal: Genome Biology (IF: 14.028) 研究背景 虽然多细胞生物中的大多数细胞都携带相同的遗传信息,但在每种细胞类型中,只有一部分基因被转录。基因表达的这种分化在很大程度上取决于调控元件的激活和抑制,其中重要的组成部分是增强子。增强子可以位于距其靶基因数十kb远的位置,因增强子的染色质和DNA有明显特征,从而可以通过全基因组分析对其进行识别。此研究表明染色质特征数据的整合分析可用于预测玉米基因组中的远端候选增强子,从而为更好地理解玉米中的基因调控提供基础。 研究材料 v2玉米幼苗,v5玉米幼苗,玉米苞叶 涉及技术 DNase-seq,ChIP-seq, BS-seq,RNA-seq 研究思路 1、使用已发表的BS-seq数据确定DNA低甲基化区域和未甲基化区域。 2、使用DNase-seq鉴定染色质可及性区域。 3、使用ChIP-seq测定H3K9ac富集区域。 4、结合BS-seq,ChIP-seq,DNase-seq数据,确定增强子候选区域。 5、使用RNA-seq分析玉米V2-IST和苞叶组织间的差异表达基因,并结合增强子候选区域,确定候选增强子区域调控的靶基因。
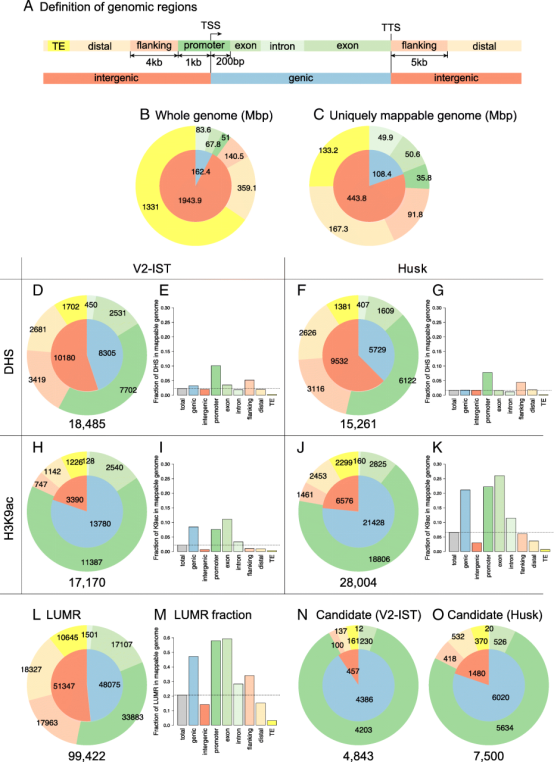
重要结果 1、鉴定潜在的顺式调控元件共有9212个基因间区的DHS(DNase I超敏位点)是潜在的顺式调控元件,通过DNase-seq绘制DHS是一种识别顺式调控元件的有效方法。V2-IST和苞叶样本中鉴定的峰之间的重叠很大,表明大多数DHS不是组织特异性的。表达水平与基因区域的DNase-seq覆盖率之间存在正相关。21,445个DHS中共有9212个(43%)位于基因间区。DHS在两种组织中占据了约2%的基因组。DHS分别占V2-IST和苞叶中启动子区域的10%和8%。 ChIP-seq识别6511个基因间区的H3K9ac富集区域。在这两种组织中,近70%的H3K9ac富集区域位于启动子;这种富集比DHS更明显,表明在没有DHS的情况下,启动子也会存在H3K9ac修饰。基因间区H3K9ac富集区总数为6511个。V2-IST和苞叶的H3K9ac富集区域分别占基因组的2%和7%。 低DNA甲基化被选为识别增强子的第三个特征,因为它与增强子活性呈正相关。使用了从B73胚芽鞘获得的公开可用的BS-seq数据,确定了CG和CHG环境中DNA甲基化为20%或更低的区域。近50%的基因区域是低甲基化的,启动子区域和外显子近60%,而几乎所有的TE都是高度甲基化的。最后确定了46,935个基因间区的LUMR作为潜在的增强子候选区域。
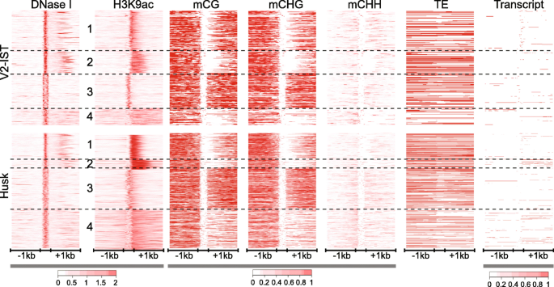
图二、 玉米基因组唯一比对部分的染色质特征分布 2、候选增强子的特征将与DHS和H3K9ac富集区域重叠的LUMR区域定义为活性候选增强子。V2-IST和苞叶中分别确定了398和1320个候选增强子,其中223个在两个组织中共享,总共产生1495个候选增强子。V2-IST中的256个和苞叶中的775个候选增强子位于5kb之外;V2-IST中的208个和苞叶中的623个候选增强子距最近的基因超过10kb。 在大多数情况下,与TE区域相重叠的候选增强子(TE-enhancer)重叠超过80%的长度或完全位于TE内。TEs与最近基因之间的平均距离在所有TEs和包含候选增强子的TEs之间没有变化(平均距离分别为40.4kb和42.5kb)。 基于H3K9ac富集模式,使用k-means聚类算法将候选增强子分为四个类别,并根据它们在热图中的展示对类别进行编号。热图显示,H3K9ac可以主要富集在DHS的一侧(1类和2类)、DHS内(3类)或存在于两侧但明显富集在其中之一(4类)。 候选增强子和TSS的DNA和染色质特征谱相似。TSS和候选增强子的热图显示染色质可及性和H3K9ac模式相似,但它们在转录水平和mCG和mCHG的分布方面存在差异。 对组织特异性候选增强子列表进行排名,1495个候选增强子被分为175个V2-IST特异候选增强子、1097个苞叶特异候选增强子和223个共享候选增强子。在玉米中,报道了五种充分表征和推定的增强子,在此研究的筛选中也识别到了b1、tb1和bx1的增强子。
图三、候选增强子的染色质、DNA和转录本特征的热图 讨论: 该论文通过对苗期叶片和苞叶的WGBS-seq,DNase-seq和H3K9ac ChIP-seq三种技术检测分别获得候选的400和1300个增强子。有三个玉米已发现的增强子在这次研究中也被发现。H3K4me1组蛋白修饰普遍认为是动物基因组的增强子标记,但是在本研究中其没有体现出增强子的特征标记特性,其主要富集于基因区。虽然LUMR区域已被认为可以用于预测增强子,但是仅有26%的区域与DNase-seq或H3K9ac ChIP-seq的富集区域重叠,有可能是因为剩下的LUMR是其他组织器官的活性增强子。 |