NEWS
新闻资讯
|
Hi-C专题(二)| 科研想提效?快来看看这个Hi-C多组学分析思路作为科研黑马的Hi-C技术,与其他组学技术(ATAC-seq、ChIP-seq、RNA-seq等)的联合分析已经成为不少高分文章的首选策略。小i同学今天结合几篇文献谈谈Hi-C多组学文章的研究思路,帮助各位老师和同学快速掌握Hi-C多组学分析的技能,挖掘染色质空间构象对基因调控的影响。 上篇Hi-C专题文章提到染色质3D结构分为4个层级:染色质疆域、染色质区室、拓扑关联结构域和染色质环。Hi-C测序后文章的分析思路也是从这几个方面展开,包括Hi-C互作热图分析,区室(compartåments)鉴定与分析,拓扑关联结构域(TADs)分析,染色质环(chromatin loops)分析。
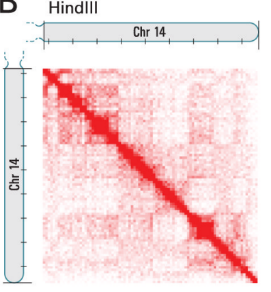
我们先从几篇Hi-C的高分文章谈谈联合分析的细节,最后再汇总联合分析的框架。 Hi-C互作图谱 Hi-C结果中最常见的是染色质接触图谱,它的横轴和纵轴表示一定分辨率的基因组坐标。下图展示的是人类14号染色体内部互作的图谱。每个像素表示一个1Mb的位点与另一个1Mb的位点之间所有的相互作用,强度对应于读取的reads数。富集到越多的reads,颜色越红。
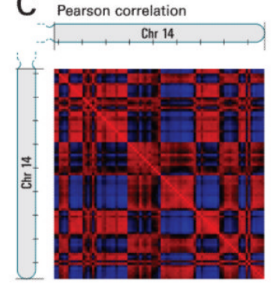
人类14号染色体互作热图 Lieberman-Aiden et al. Science. 2009 了解完染色质接触图谱后,我们再来关注相关矩阵图。下图仍然是人类14号染色体,该图表示每对1Mb基因座互相作用的相关性热图。相关性计算数据可以将染色体分割成两部分(红色和蓝色部分)。
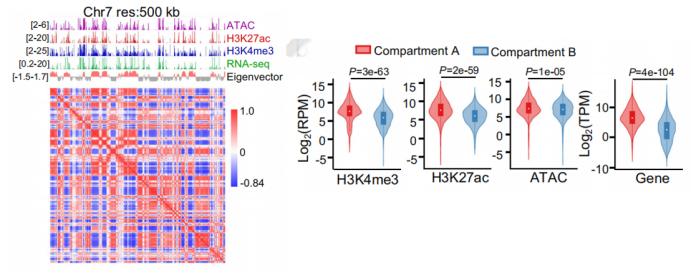
人类14号染色体内部互作(1M位点)相关性热图 Lieberman-Aiden et al. Science. 2009 下图来自论文“A compendium and comparative epigenomics analysis of cis-regulatory elements in the pig genome”,作者对大白猪(LW)的肌肉组织7号染色体进行ATAC-seq、 ChIP-seq和RNA-seq信号富集分析,同时联合Hi-C相关性热图。从图上可以明显地看出染色体分为活跃的A compartments和不活跃的B compartments。在A compartments对应位点区域有更多的ATAC、H3K27ac、H3K4me3和RNA-seq信号富集,联合这些信息方便后续从中挖掘受到调控的基因及其调控的潜在机制。
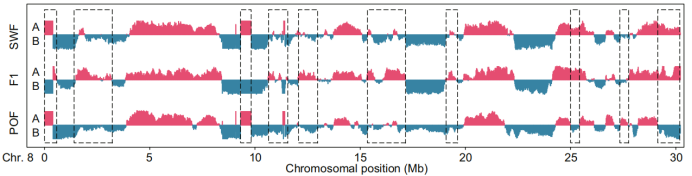
500kb分辨率下LW猪肌肉组织7号染色体的ATAC-seq、ChIP-seq、RNA-seq富集信号与Hi-C相关性图谱 Zhao et al. Nature communications. 2021 Compartments的鉴定与分析 上文提到相关矩阵图可以将染色质分为两类,对应于compartment A和compartment B。原理是用主成分分析法对每条染色体进行了分割,将染色体的PC1向量与基因组特征(基因密度与GC含量,或只有基因密度)进行皮尔森相关系数计算,正值为compartment A,其余为compartment B。分析时关注全基因组A/B区室保守区及A/B区室转换区(比较不同时期、不同组织、不同品种),重点分析转换的区室。 下图来自“Dynamic transcriptome and chromatin architecture in granulosa cells during chicken folliculogenesis”一文,作者研究了鸡卵泡发育过程中几个时期(3个关键发育时期:SWF、F1和POF)的染色质结构和转录变化。从图上可以看出8号染色体一些位点区域在3个时期存在明显的区室转换(虚线区域),意味着这些区域有些基因转录状态从活跃到关闭或从关闭到活跃,相应地基因的表达水平可能会发生变化。
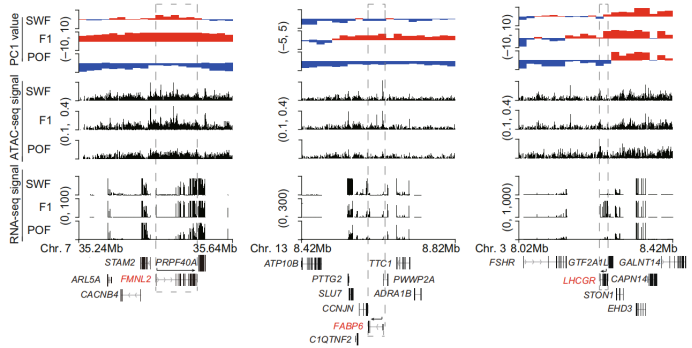
8号染色体区室分割图,红色代表compartment A,蓝色代表compartment B Li et al. Nature Communications. 2022 与RNA-seq做联合分析时可以统计分析A/B转换区域的基因数量、功能富集和表达水平,从而确定哪些基因的表达水平受到染色质结构变化的调控。最后,筛选到调控生物功能的候选基因。与ATAC-seq联合分析时,分析发生A/B区室转换位置的ATAC-seq信号,分析其中特异的peaks。 下图也选自鸡卵泡发育的研究论文,对区室转换中的基因进行功能富集分析。三个代表性基因(FMNL2、FABP6和LHCGR)选出来进行区室转换、ATAC-seq信号和RNA-seq信号比较分析,可以看出它们在3个时期或多或少存在区室转换的变化、ATAC-seq信号以及RNA-seq信号的差异(虚线框框)。
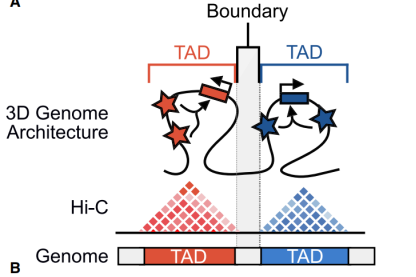
三个发育时间A/B区室转换区域的ATAC-seq信号和RNA-seq信号比较分析 Li et al. Nature Communications. 2022 拓扑关联结构域TADs分析 拓扑关联结构域TADs是继染色质区室更加精细的结构,TADs主要是由一些高度互作的区域组成,边界是一些低互相作用区域。边界主要由一些绝缘元件(比如绝缘子蛋白和黏连蛋白)组成,限制顺式作用序列的互作。TAD边界富集管家基因和转录起始位点,在培养的细胞和体内移除绝缘的TAD边界会导致基因的异位表达。TADs分析时主要关注拓扑结构域内部和边界(boundary)的变化。 TADs分析图一般展现为三角形热图(下图),每个小三角形代表着一个TAD。染色质结构的改变可能会导致TADs的变化(尤其是边界的变化),在研究的过程中我们一般会关注不同样本间TADs边界的变化。
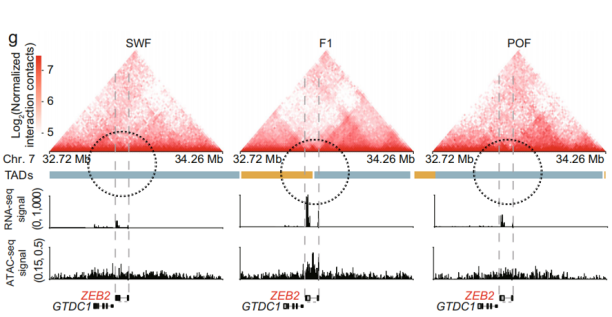
McArthur E and Capra JA. Am J Hum Genet. 2021 下图选自“Dynamic transcriptome and chromatin architecture in granulosa cells during chicken folliculogenesis”,展示了基因ZEB2位点(chr7:32.72-34.26Mb)TAD边界的变化,联合RNA-seq和ATAC-seq信号分析。 基因ZEB2可以调节细胞-细胞粘附和细胞骨架结构重排,F1时期出现了一个明显的TAD边界(虚线),正是ZEB2基因所在的位置,联合分析表明ZEB2基因的表达水平在F1时期显著提高,另ATAC-seq信号在F1时期也呈现出明显的峰。
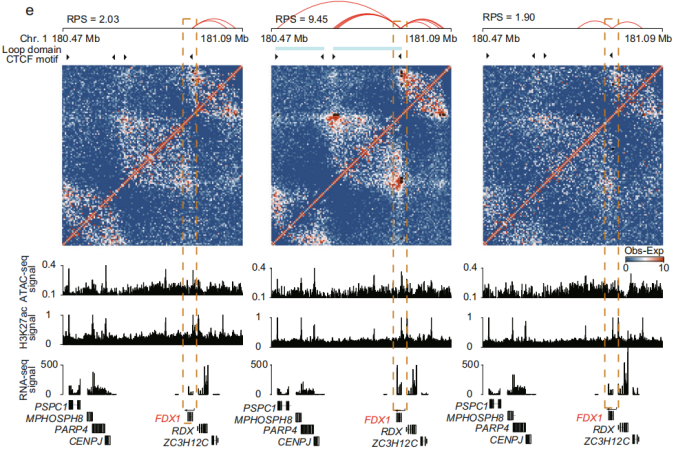
不同发育时期ZEB2基因位点TAD边界及表达水平的变化 Li et al. Nature Communications. 2022 TAD边界通常与组蛋白修饰、甲基化修饰等有关,所以各位老师可以借助Hi-C与ChIP-seq、ATAC-seq、RNA-seq等组学技术联合分析找到TAD变化区域的基因以及它们表达调控的变化。这是一个研究切入点,尤其是那些关注时空差异、组织特异或者品种特异的样本。 染色质环chromatin loops 染色质环经常连接启动子与增强子,与基因的激活有关,因此PEIs(promoter-enhancer interactions)的鉴定与分析也是关注的热点。此外,上述研究显示不同的样本存在着区室和TADs边界的变化,但不少研究结果显示大部分的A/B区室、拓扑关联结构域在不同的细胞类型、品种中是保守的。PEIs在组织和发育时期更具动态变化,故在3D基因组的研究中关注PEIs的变化会更有针对性。 对于PEIs的分析会调用一个调控潜力分数(regulatory poteintial score,RPS),一般RPS值的增加与基因表达的增加有关。下图是鸡卵泡文章的PEIs重连分析图(基因FDX1基因坐标区域的启动子-增强子互作连接图和Hi-C互作热图),以及同坐标位置的ATAC-seq、H3K27ac、RNA-seq信号图。 基因FDX1与甾类激素的合成有关,结果显示它的PEIs在不同时期发生了重连变化,F1时期(中间图片)尤其值得注意,RPS值最大,PEIs的数量(Hi-C互作热图的黑点代表PEIs)也最多。联合分析显示FDX1的RNA-seq信号在F1时期明显增加。H3K27ac的分布可用于测量PEIs中潜在的增强子活性,该研究暗示在F1时期FDX1受到超级增强子的影响。
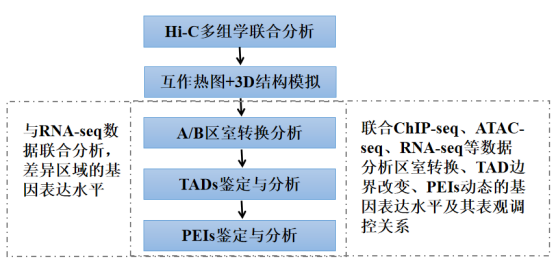
功能基因FDX1 基因区域PEIs和Hi-C接触图谱 Li et al. Nature Communications. 2022 Hi-C多组学联合分析框架 Hi-C技术可以用于辅助基因组组装,也可以用于比较分析不同样本的三维结构变化。联合ChIP-seq、ATAC-seq、RNA-seq等组学技术分析基因表达与调控变化的研究思路已经相对成熟。联合分析的框架大致上如下,可以单独联合RNA-seq数据分析染色质结构变化区域的基因表达水平的差异,也可以联合ChIP-seq、ATAC-seq、RNA-seq数据挖掘染色质结构改变对相应位置基因表达水平、转录调控的影响。
结语: 不同品种或同一物种不同组织、不同发育时期或逆境胁迫下,染色质的3D结构存在或多或少的动态变化,预示着其对基因的表达可能有潜在的调控作用。利用Hi-C挖掘染色质3D结构的改变是研究基因表达调控的有利工具,相关领域的老师和同学,不要犹豫赶紧上车吧! 参考文献: 1.Lieberman-Aiden et al. Comprehensive mapping of long range interactions reveals folding principles of the human genome. Science. 2009. 326: 289-93. 2.Li et al. Dynamic transcriptome and chromatin architecture in granulosa cells during chicken folliculogenesis. Nat Commun. 2022. 13:131. 3.Zhao et al. A compendium and comparative epigenomics analysis of cis-regulatory elements in the pig genome. Nature commun. 2021. 12:2217. 4.McArthur E and Capra JA. Topologically associating domain boundaries that are stable across diverse cell types are evolutionarily constrained and enriched for heritability. Am J Hum Genet. 2021. 108:269-283. |