NEWS
新闻资讯
|
项目文章 | 转录组和small RNA测序联合分析揭示矮牵牛花形态发育的动态调控机制
发表单位:中南林业科技大学园林学院 发表日期:2022年3月28日 期 刊:horticulturae(IF: 2.923) 研究材料:矮牵牛花 2022年中南林业科技大学园林学院文亚峰教授团队在horticulturae(IF: 2.923)期刊发表题为“An Integrated Analysis of Transcriptome and miRNA Sequencing Provides Insights into the Dynamic Regulations during Flower Morphogenesis in Petunia”的研究论文。该研究利用转录组和miRNA测序技术,捕捉了矮牵牛花在五个不同发育阶段的动态表达模式。研究结果为由转录因子、miRNA及其靶基因促进发育基因调控网络提供了新见解。爱基百客为该研究提供RNA-seq和small RNA Sequencing技术支持。
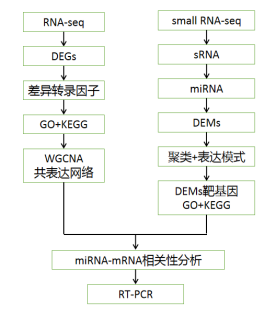
01 研究背景 花是植物中最复杂的结构,探索其形成的调控机制在进化和生殖生物学中具有重要意义。迄今为止,研究发现花的形成涉及不同的发育过程,如花分生组织命运的诱导、花器官特性的确定、花器官的形态建成和成熟等。除了转录水平的遗传机制外,表观遗传调控因子,尤其是microRNAs,参与了花发育的许多方面。因此,该研究以转录组和miRNA测序为工具分析了花形态建成过程中的调控机制。 02 研究思路
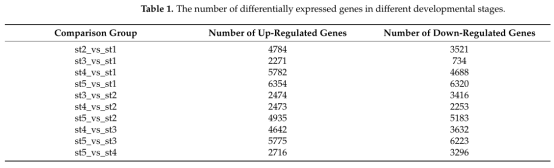
03 研究结果 01 矮牵牛花发育过程中转录组的全局变化 选取矮牵牛花盛开前5个发育阶段的花蕾(花蕾5cm)进行转录组测序。测序完后获得723,670,362个clean reads,并对不同时期的差异表达基因统计分析(表1)。
图1 矮牵牛不同花期的研究图像
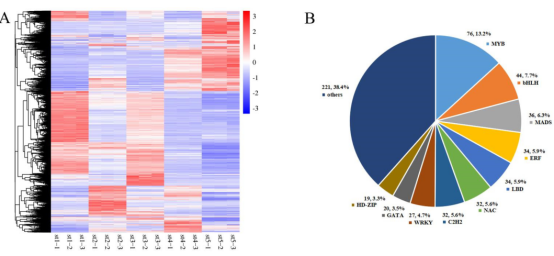
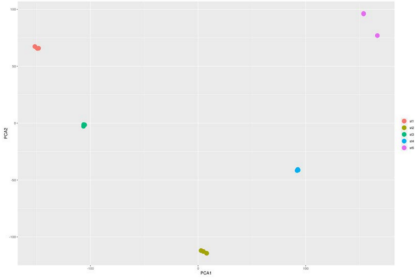
表1 不同发育阶段差异表达基因的数量 图2A显示了所有样本的全局聚类和显著的基因表达变化,在这些样本中可以观察到花发育过程中一个阶段特异性的转录组谱。同时,大量下调的DEGs在st4和st5中表现出相似的表达模式。主成分分析(图S1)结果表明样本可以区分开,3次重复的样本聚在一起。
图2 差异表达基因汇总
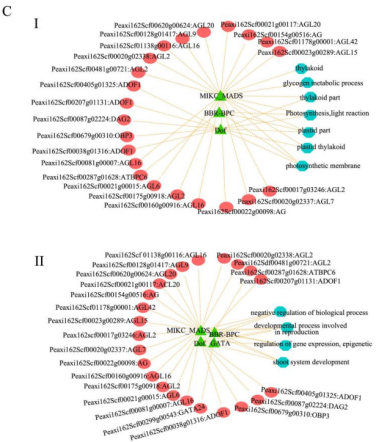
图S1 所有样本的主成分分析 02 差异表达转录因子 转录因子基因在花的发育过程中起着至关重要的作用,并且许多属于不同转录因子家族的成员已经被证实在花形成的各个方面都很活跃。FDR<0.05以及|logFC|>2为标准筛选,共有575个DEGs编码转录因子,并且根据转录因子数据库将它们划分到39个TF家族。占比最多的前10 TF家族在图2B中展示,MYB基因家族占比最多(76;13.2%),其次是bHLH、MADs、ERF和LBD。TF家族中表达差异最大的TF基因都出现在st5和st1中,对应于形态差异最大的时间段。 03 DEGs的GO富集和KEGG通路分析 对2个不同时期的花的差异基因(DEGs)进行GO和KEGG富集分析。作者重点分析了其中上调和下调基因的富集GO项,比如上调DEGs中显著富集的GO项包括“蛋白激酶活性”(GO:0004672)、“蛋白磷酸化”(GO:0006468)和“转移酶活性、转移含磷基团”(GO:0016772)存在于st4 vs. st2,st5 vs. st2, st5 vs. st3和st5 vs. st4。 KEGG富集分析结果显示整体上矮牵牛花形态发育过程中碳水化合物代谢呈增加趋势,光合作用呈减少趋势。此外,富集通路涉及苯丙氨酸代谢,包括类黄酮生物合成、苯丙素生物合成和苯丙氨酸代谢,同时发生在st2 vs. st1、st4 vs. st1、st5 vs. st1、 st4 vs. st2、st4 vs. st3、st5 vs. st3的上调的DEGs中。次生代谢产物在花器官分化和生长过程中起着重要作用。 04 基因聚类分析和共表达网络构建 根据基因在不同发育阶段的表达模式,将具有相似转录组谱的基因聚类。共鉴定到9个不同的表达集群,通过WGCNA分析进一步分为6个模块:蓝色、棕色、绿色、红色、绿松石色和黄色模块(图3A)。在每个模块中,基因在表达模式上具有较高的协同性。作者对每个模块的共表达基因的表达模式进行分析,还对基因进行GO和KEGG功能分析(图3B)。 根据WGCNA结果,作者选择蓝色和yel+tur(黄色和绿松石色的基因表达模式基本相似,作者将这两个模块的基因作为一个样本)模块中的基因构建一个假定转录调控网络,将启动子区含有结合motif的共表达基因与具有结合位点的转录因子基因连接起来。如图3C(I)所示,蓝色模块显示MIKC-MADS、BBR-BPC和Dof的DNA序列基序高度富集,主要与AGL2、AGL16、AG、AGL9、ATBPC6、ADOF1、DAG2和OBP3等转录因子相关。靶基因在类囊体、糖原代谢过程、类囊体部分、光合作用、光反应、质体部分、质体类囊体和光合膜等方面富集GO。MIKC_MADS、BBR-BPC、Dof和GATA的DNA基序在图3C(II)的yel+tur模块中发现,TFs包括AGL9、AGL20、AGL16、ATBPC6、ADOF1、OBP3和GATA24。该模块的靶基因涉及GO项的生物过程负调控、生殖发育过程、基因表达调控、表观遗传和茎系统发育。
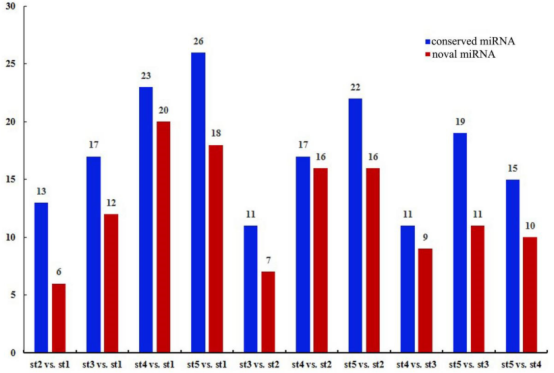
图3基因聚类分析与共表达网络构建 05 矮牵牛的sRNAs分析 对15个文库进行small RNA-sequencing,产生了9,838,267-22,586,531的原始reads。对clean reads进行注释,去除rRNA、snRNA、snoRNA和tRNA等非编码sRNAs,大约74.92%的clean reads成功与矮牵牛花参考基因比对上。作者分析了至少两个不同样本的总和unique sRNAs的分布,在st5 vs. st1中鉴定到最多的总sRNA,st2 vs. st1鉴定到最多的unique sRNA。大多数sRNA的长度为21-24nt;24nt是最大的一类,21和22nt 类在研究的样本中比23nt类更丰富。 利用miRbase 17.0鉴定到45个保守的miRNAs,包括miR156、miR159、miR160、miR164、miR166/165、 miR167、miR172和miR319 位点。每个发现的miRNA赋予一个ID,格式:Ph-miR(number)。在miRNAs显示与miRBase序列相同的情况下,使用注释Ph-miR (number)/miRBase。最常见的是Pa-miR36/stu-miR395j(12loci)和Pa-miR29/stu-miR166d-3p(8loci),这些miRNAs的前体长度在41-252nt之间。基于茎环结构特征,预测到26个新miRNAs。对发现的miRNA进行表达模式分析,结果暗示miRNA表达表现出随时期或组织而异的特异性,这与红瓜(Coccinia grandis)的性别特异性miRNA在性别分化的激活起重要作用以及拟南芥花同位异型转录因子和miRNA调控花发育的动态调控网络中类似。 06 花发育过程中miRNAs的差异表达 以padj<0.05和|log2FoldChange|>1为标准筛选两个不同发育阶段间显著差异表达的miRNA(DEMs)。在st5 vs. st1中鉴定得到最多的44个DEMs,其包括26个已知的miRNAs(9个下调,17个上调)和18个新的miRNAs(10个下调和8个上调)。作者绘制了一个热图来描述所有DEMs的聚类和整体表达模式,显示出明显的上调miRNAs的阶段特异性表达。
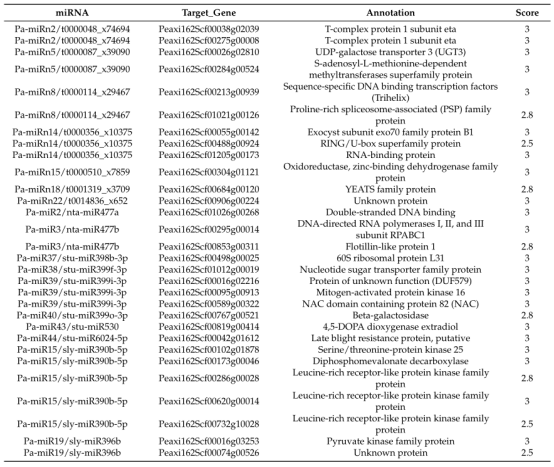
图4 两个不同发育阶段差异表达miRNAs的数量 07 DEMs靶标的GO富集和KEGG分析 利用psRobot软件预测保守和新的miRNAs的潜在靶标,选择两个不同时期的靶标DEMs进行GO功能富集和KEGG通路富集分析,以q-value<0.05作为差异阈值。DEMs的靶基因在st2 vs. st1、st3 vs. st1、 st4 vs. st1和st5 vs. st1全富集在响应激素反应(GO:0009725)的GO项,包括ARF6、ARF10和ARF8,它们可能被miR160和miR167调控。显著富集的KEGG通路分析仅在st2 vs. st1、st4 vs. st2、st5 vs. st2、st4 vs. st3、st5 vs. st3五个比较组中进行。 08 矮牵牛花发育不同阶段miRNA-mRNA的相关性分析 根据保守miRNA和新miRNA靶基因预测结果,基于5个不同发育阶段样品的表达水平,分析miRNA和mRNA测序谱之间的相关性,筛选相关性>0.8负相关的miRNA和靶基因对,构建矮牵牛花发育过程中miRNA - mRNA调控网络。
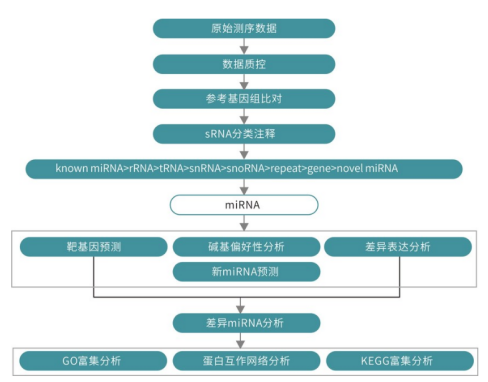
表2 miRNA与其靶基因的相关性分析 09 定量RT-PCR验证差异表达基因和miRNAs 选出9个DEGs和9个DEMs进行RT-PCR,对转录组和miRNA测序结果进行验证。结果显示大部分基因或miRNA在5个不同发育时期表现出相似的表达模式,而其中少数在两个相邻的阶段表现出对比的趋势;这可能与测序技术和qRT-PCR分析差异有关。 04 研究结论 该研究利用RNA-seq和miRNA测序全面描述了矮牵牛5个花发育阶段的mRNA和miRNA的动态表达谱。大量的候选基因和miRNAs被鉴定出来,表现出时间表达模式的特征。在表达基因中观察到共表达模式,并提示存在新的miRNA-mRNA通路。对DEGs和DEMs的靶基因进行富集分析表明碳水化合物代谢、光合作用、苯丙氨酸代谢和植物激素途径在花发育过程中起着重要作用。这些结果将有助于进一步研究转录因子的特异性、不同发育阶段的基因调控机制以及花形态差异的遗传基础。 Small RNA-seq产品介绍 small RNA 是生物体内一类重要的功能分子,广泛存在于高等和低等生物体内。Small RNA 与细胞的发育、分化、疾病等密切相关。近年来的研究表明,small RNA 不仅可作为癌症诊断、识别、预后的标志物,也在癌症治疗中发挥作用。 small RNA包括microRNA、ncRNA、siRNA、snoRNA、piRNA 等。它们的主要功能是诱导基因沉默,调控细胞生长、发育、基因转录和翻译等生物学过程。通过mRNA 降解、翻译抑制、异染色体形成以及DNA 去除等多种途径,广泛地参与调控生物体的生长发育和疾病发生。 基于新一代高通量测序技术的small RNA 测序,可一次性获得数百万条small RNA序列,能够快速地鉴定某种组织在特定状态下的所有已知small RNA 并发现新的small RNA,以及预测small RNA 的靶基因,为small RNA 功能研究和调控机制提供有力的工具。 技术路线
有small RNA-seq需求的老师欢迎和我们沟通! |