NEWS
新闻资讯
|
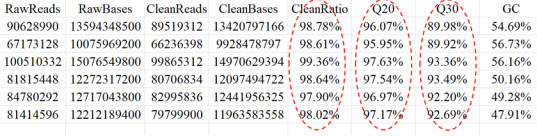
转录组专题 | 保姆级教程,手把手教你看懂真核转录组测序结果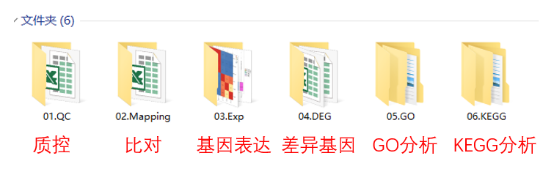 图1. 转录组结果文件夹展示 一、数据的质控 拿到分析结果后,我们首先看到的是原始数据整理与质量评估,数据量的大小与测序质量的好坏是评判测序数据可靠性的重要标准,是保证可以得到理想结果的必要条件。一般原始数据用Raw data来表示,对于绝大部分真核生物来说,转录组测序6G数据量即可,若老师需要获得更多低丰度基因的信息,测序数据量可达到10G甚至更多。 原始数据通常需要过滤得到Clean Data,Clean Data的数据评估中主要包括碱基质量、碱基分布以及碱基含量。图2是数据统计表,其中Raw reads和Clean reads分别代表原始测序 reads 数和过滤得到的 reads 数,Raw Bases 和Clean reads分别代表原始数据的总碱基数和过滤后的总碱基,CleanRatio则代表 clean reads 所占比例,理论上越高越好。 在Clean Data质量分析中我们通常会看到Q20和Q30这两个概念(类似于产品合格率),它们分别代表碱基错误识别率为1% 和0.1%。我们通常选择Q30作为碱基质量评价标准,Q30的值越大越好,一般大于85%结果会比较好,对应到图3a整体碱基质量分数在绿色区域(>28)是比较好的结果。碱基含量即ATGC四种碱基所占的比例,一般除了前一小段碱基位置之外(大概10bp左右,这是二代测序的通病),4种碱基的含量线条应呈现出平行且接近的状态(图3b)。此外,在测序过程中识别不了的碱基(N碱基)的含量越少越好,一般接近于0(图3c)。
图2.数据统计表 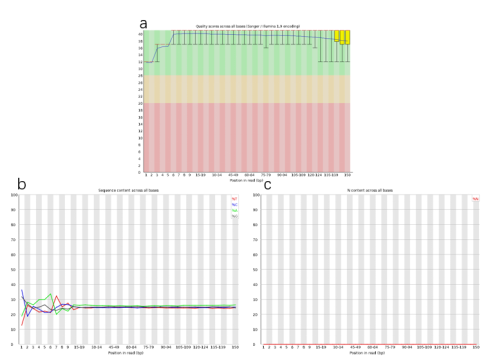 图3.数据评估结果展示 二、数据比对与定量 接下来,我们看数据分析结果,这部分包括基因组比对分析、表达定量分析、PCA 分析、样本间相关性分析、reads在基因功能元件上分布、差异表达分析、差异基因的GO分析以及 KEGG 分析。其中差异基因的鉴定与功能富集分析是转录组文章的重点内容,数据挖掘与分析也是基于这两个模块进行。
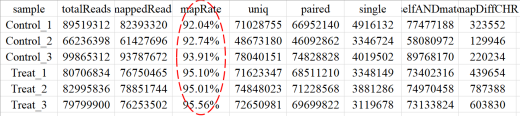
图4.比对结果统计 在这一部分,我们首先来看基因组比对分析,就是将 Clean reads 数据使用 hisat2 软件 (version: 2.0.1-beta) 比对到参考基因组,比对结果会出现如图4所示,在表中我们需要关注mapRate,除了样本污染的非常规因素,比对率主要与参考基因组与目标物种的适配度有关,参考基因组的选择也是转录组分析比较重要的影响因素(详情可参考爱基百客公众号《如何填写测序项目信息分析表》)。
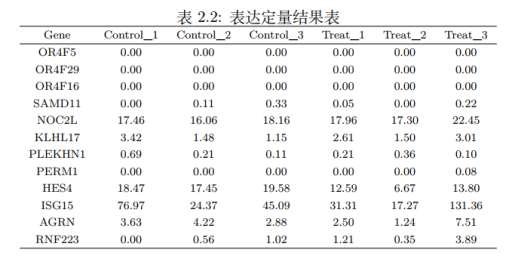
接下来,我们来看基因表达定量分析。在得到有效 reads 之后,我们使用 featureCounts软件 (version: v1.6.0) 根据基因组的注释文件统计出 reads 落在基因上的数目,由于每个样本的测序量不一样,为了能横向比较同一基因不同样本间的表达量差异,我们需要对基因的 reads 数进行标准化,这里采用的标准化方式为FPKM,代表每千个碱基的转录每百万映射读取的fragments,主要是针对pair-end测序表达量进行计算。其实,我们通常还会看到另一种标准化的表达方式叫RPKM(R代表reads),这其实跟FPKM类似,它们的区别就在于FPKM适用于双端测序文库,RPKM适用于单端测序文库。
FPKM实际上是一个相对定量,通过横向对比可以得出不同样品之间同一基因表达量的差异。
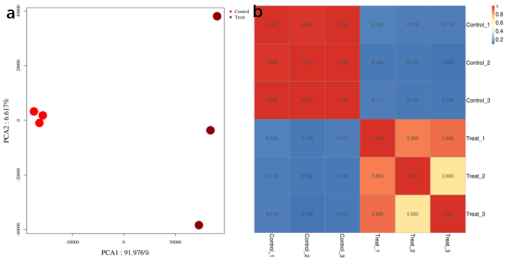
图6. PCA 分析和样本间相关性分析 生物学重复之间的相关性高低与差异基因鉴定的准确性息息相关,我们通常采用两种算法进行重复性的评估(图6)。主成分分析(PCA)是设法将原来众多具有一定相关性的指标(如reads 的分布特征),重新组合成一组新的互相无关的综合指标,从而降低问题的复杂性,来研究样品间的主成分关系。二维PCA图将主成分 1(PC1)和主成分 2(PC2)分别作为 X 轴和 Y轴的散点图,图内每个点代表 1 个样本。如果两个样本距离越远,则说明两个样本 reads分布的差异越大。反之,则说明相应样本 reads 整体分布模式越接近。理想情况下,生物学重复的样本应该聚类在一起,而处理组间应该可以清晰区分开(图6a)。除PCA外, pearson相关性分析热图也是用于衡量生物学重复的方法,热图中会展示相关性具体数值,并用不同的颜色进行标注,相关性越高颜色越深。正常情况下,组内重复的相关性最好是大于不同组之间的相关性。
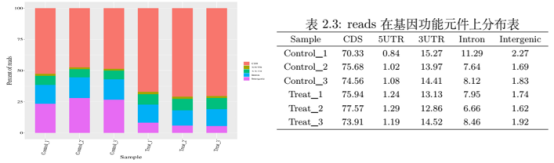
图7. reads 在基因功能元件上分布 同时我们将比对到基因组上的有效 reads 按照功能元件进行统计,把基因组分为CDS、 5UTR、 3UTR、 Intron 和 Intergenic 区域分别统计落在其上的 reads 比例。正常情况下, RNA-seq 的reads 大部分会落在 CDS 区域,统计结果如图7。
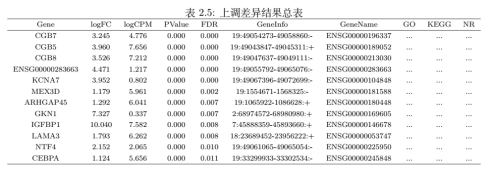
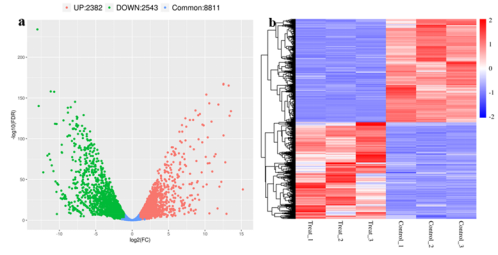
图8. 差异表达分析 三、差异基因分析与富集分析 最后,来到我们转录组测序结果最核心的部分,差异基因的鉴定与功能富集分析。首先,为了比较不同样本间的基因表达量差异,我们通过R包edgeR进行差异表达分析,筛选阈值设定为FDR < 0.05&| FoldChange | > 2 ,结果如上表2.5所示,FDR为错误发现率,一定程度上对p值的假阳性进行了校正,而FoldChange则表示差异倍数,在文章或结果中常见以log2FC进行展示。差异基因的可视化一般以火山图进行展示(图8a),绿色代表下调基因,红色代表上调基因,对FDR取了负log,负log就越大,两个基因之间的差异也越显著,所以在火山图里,越外层的岩浆差异越大,火山图可以更加形象的展示出整体表达差异信息。图8b 代表差异基因heatmap图,这张图中的红色和蓝色分别代表基因的上调和下调,颜色的深浅代表基因的上下调程度,通过差异基因heatmap图我们可以得到不同处理样品之间基因表达的差异。
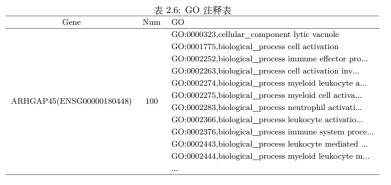
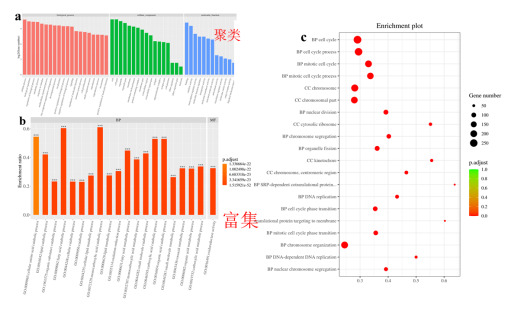
图9. 差异基因的 GO分析 将GO注释对应的gene数目,进行统计然后按照分子功能 (Molecular Function)、细胞组分 (Cellular Component) 和生物学过程 (Biological Process) 分类绘图,结果如图9a所示,基因可以通过ID对应或者序列注释的方法找到与之对应的GO号,而GO号可对应到具体的term,即功能类别或者细胞定位,这也是GO富集的一个基础。而富集的目的就是从差异显著的基因中挑选出我们需要的基因,并分析挑选结果是否可信。GO 富集柱状图和富集气泡图(图9b、c),它们都是GO 富集结果直观展示。气泡图气泡的大小代表基因的数目,颜色代表P值,P值越小基因数目越多代表我们得到的结果越可信。总之,通过差异基因的GO 分析,可以找到富集差异基因的GO分类条目,从而寻找不同样品的差异基因可能与哪些基因功能的改变有关。
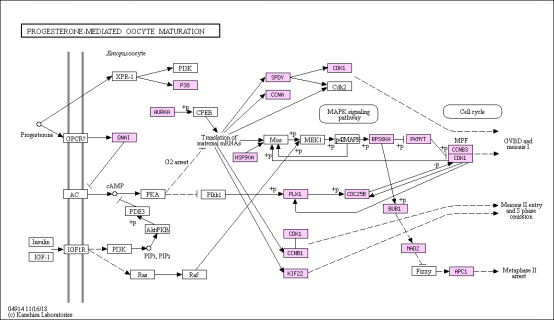
图11. KEGG代谢通路富集分析 通路分析:Pathway是指在系统水平上完成生物的某一功能的基本单元或者局部子网络,这其中的 Pathway是内容是 KEGG 的核心。目前针对 Pathway 的分析、注释,大多数是基于 KEGG-Pathway 来做的。气泡图是 KEGG 富集分析结果的图形化展示方式。在此图中,KEGG 富集程度通过 Rh因子值和富集到此通路上的基因个数来衡量。此外,用P <0.05筛选出显著富集的通路,进而查找与研究相关的生物学途径。在此代谢通路图中,差异基因映射的位置会用彩色标示出来(图11)。总之, GO和KEGG就是两个数据库,它们里面有每个基因相关的功能信息,通过注释和富集分析就是把这些功能信息进行整合计算的一种算法。 好了,今天就了解到这里吧,之后我们还会持续分享测序的相关知识点,持续关注哦。爱基百客生物专注表观和单细胞测序技术服务,各种组学实验都有丰富的经验,有相关需求的老师可以联系我们。 |