NEWS
新闻资讯
|
经典综述解读 | 转录组测序的这些年(上)转录组学始于上个世纪90年代,经过几十年的发展,已经成为生物研究不可或缺的工具之一。技术的革新让转录组的应用范围不再局限于基因表达分析,爱基百客旗下除了常规RNA-seq,还有LncRNA-seq、Small RNA-seq、全长转录组等产品。这些转录调控的产品帮助许多老师解决了科研上的问题。本期我们和大家分享一篇转录组的经典综述,让大家全面了解转录组学及其发展历史,是一篇值得收藏的综述。
在过去10年时间里,RNA测序(RNA-seq)已成为差异基因表达和可变性剪接分析不可或缺的工具。然而,随着测序技术的高速发展,RNA-seq技术也在不断革新。现在,RNA-seq技术可用于研究RNA生物学的许多方面,包括单细胞转录组、翻译(翻译组)、RNA结构(结构组)以及空间转录组。将新的长读长(long-Reads)和长链RNA直接测序(direct RNA-seq)技术与数据分析计算工具相结合有助于我们更加全面的理解RNA生物学过程,例如转录从何时、何地发生再到控制RNA功能的折叠和分子间相互作用等问题。 前言:RNA测序(RNA-seq)是在十多年前发展起来的一种测序技术,从那时起,它已经成为分子生物学中无处不在的工具,RNA-seq最常用于分析差异基因表达(DGE)。RNA-seq整个实验流程步骤如下: 1. RNA提取; 2. mRNA富集或者核糖体RNA(rRNA)去除; 3. cDNA合成以及文库制备; 4. 高通量平台(通常为Illumina)上对文库进行测序,每个样本测序深度为10-30M reads(reads就是测序读出一条序列); 5. 对测序数据分析:将测序得到的Reads比对到基因组或者重新组装成转录组,对覆盖在转录组上的Reads数进行量化和标准化,通过阈值筛选出两样本间存在显著性差异表达的基因。 早期RNA-seq数据主要来源于组织块,广泛应用于各种生物体中,例如拟南芥、酵母、玉米、小鼠以及人类。虽然,RNA-seq这个术语包含了不同的方法和生物学应用,但是DGE分析仍然是RNA-seq最主要的应用(表1)。
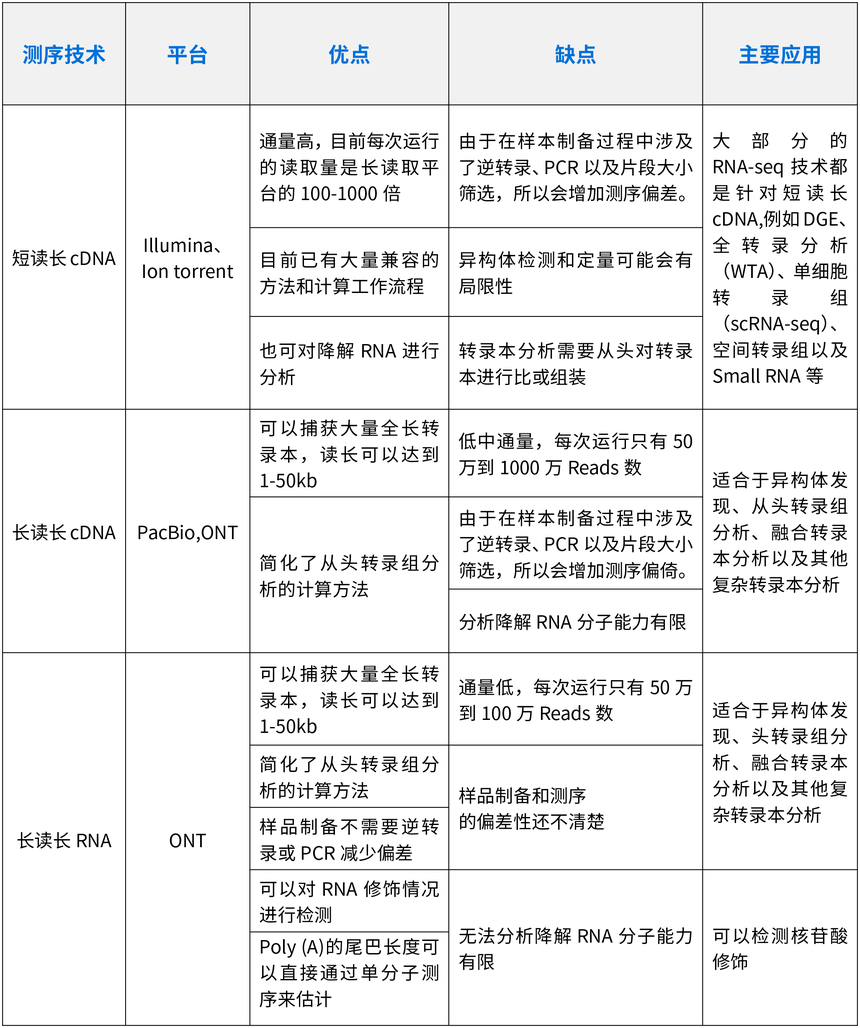
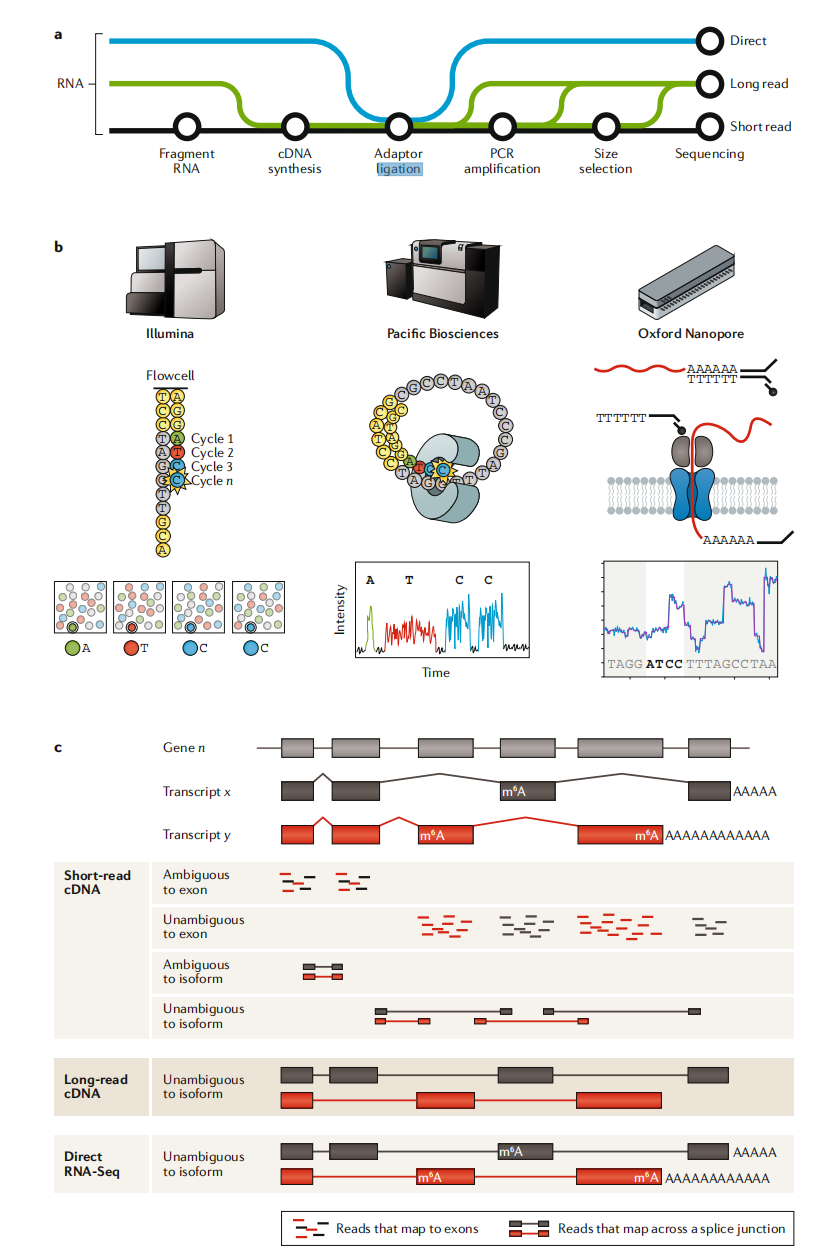
表1短读长和长读长RNA-seq平台比较 RNA-seq对mRNA剪接程度、非编码RNA相关分析加深了我们对生物学许多方面的理解。RNA-seq技术随着实验方法和计算机发展也在不断更新和完善,与以前基于微阵列的方法相比,RNA-seq提供了更为丰富的转录组相关信息。到目前为止,已经从标准的RNA-seq方法中衍生出了近100种不同的方法。即便大部分短读长cDNA文库都可以采用Illumina平台进行测序,但是仍有一些生物学问题无法利用短读长测序解决,因此长读长(long-Reads)和直接RNA-seq测序(direct RNA sequencing, dRNA-seq)应运而生。 Illumina短序列读长测序技术为SRA数据库提供了95%的转录组测序数据。由于cDNA短读长测序包含了几乎所有公开可获得的mRNA-seq数据,因此我们认为这是一种最基础的 RNA-seq技术,接下来我们会讨论关于此技术的主要流程以及局限性。随着科研学者对于转录本信息丰富度要求越来越高,长读长cDNA测序和dRNA-seq将来在转录组测序中可能会处于主导地位(图1,表1)。
短读长测序已经成了在整个转录组范围内对基因进行检测和定量的常用方法,部分原因是它比微阵列更便宜、更容易实现,但主要是因为它生成了全面、高质量的数据,可以得到整个转录组中基因的表达水平。使用Illumina平台完成短读长测序后,DGE分析的核心步骤包括:RNA提取、cDNA合成、接头连接、PCR扩增、测序和数据分析(图1)。 由于在短读长测序实验流程中存在RNA片段化、长度选择和文库纯化这些操作,所以最终的cDNA片段通常都是小于200bp。每个样本会获得20-30M Reads数,在经过数据处理后,通过这些Reads对基因表达水平进行定量,随后通过统计学方法筛选出其中存在显著性差异的基因。因为短读长测序方法比较成熟和稳定,因此在不同平台间兼容性也较为可观。但是,由于短读长测序在样本制备过程和数据分析中环节较多,所以不可避免的出现一些偏差。正是因为这样的局限因素,可能会导致在分析异构体过程中无法准确的判断基因来源,尤其是对于那些长度较长,可变度较高的异构体来说,这样偏差将会进一步放大。尽管短读长RNA-seq可以对最长的转录本进行详细的分析,但是所使用的实验方法并不能扩展到全转录组分析。其他的偏差与局限来源于那些大量的计算方法,例如如何处理模糊或多重比对Reads。 虽然Illumina在RNA-seq测序平台中占主导地位,但是Pacific Biosciences(PacBio)和Oxford Nanopore(ONT)公司都提供了可选择的长读长技术,能够对完整的单个RNA分子在转换成cDNA后进行单分子水平级的测序。整个过程中避免了短RNA-seq测序数据组装的过程,也克服了一些短RNA-seq测序相关问题。例如减少了序列读取映射中的模糊性、可以识别更长的转录本获取更完整的异构体多样性信息、降低了短读长RNA-seq中剪切体分析的假阳性率。 PacBio的Iso-Seq技术可以对长度为15kb全长cDNA进行测序,帮助我们发现新的转录本,同时还可以检测跨物种的全长同源序列来进行早期基因预测。在标准的Iso-Seq操作流程中,通过使用一种模板转换逆转录酶将高质量RNA逆转录成全长cDNA,然后再通过PCR进行扩增,扩增后的产物用作PacBio单分子实时(SMRT)文库的制备。由于较短转录本可以快速扩散到测序芯片活性表面,而且测序过程中会出现偏差,所以片段选择一般建议在1-4kb范围内。PacBio测序对于模板需求量较大,所以需要进行多轮PCR,这个过程还需要后续完善,以减少过度扩增所带了的影响。扩增得到的cDNA进行末端修复和PacBio SMRT接头连接后,就可以进行长读长测序;通过改变测序芯片的加载条件,可以进一步控制片段大小。 ONT cDNA测序方法也能产生全长的转录本,甚至还可以在单细胞水平上进行读长,同样是以模板转录逆转录酶制备全长cDNA,通过PCR进行选择性扩增,然后连接接头构建文库进行测序。直接cDNA测序会消除PCR带来的偏差,从而提高测序结果的质量,但是,PCR扩增的cDNA文库的测序产量会更高,客户所需要的RNA起始量会更少。这两种长读长cDNA方法都受限于标准模板逆转录酶的使用,这种逆转录酶可以将全长RNA和截短的RNA逆转录成cDNA。逆转录酶只会将含有5ʹ帽子结构的mRNA转换为cDNA,避免了降解后或者剪接不完全的RNA合成cDNA,从而提高测序数据质量。但是,有证据表明逆转录酶会对ONT平台的读长产生负面影响。 上述提到的长读长cDNA测序,这种方法与短读长测序平台一样,它们都需要将mRNA转换为cDNA。Oxford Nanopore公司通过实验证明他们的纳米孔测序技术可以直接使用RNA进行测序,换句话说,这种测序技术不再需要cDNA合成和PCR扩增步骤。这种方法被称为dRNA-seq,消除了之前测序技术所存在的偏差问题,并使表观遗传信息被保留。RNA的文库制备涉及到两个接头的顺序连接。首先,一个带有寡聚物(dT)的双链接头退火并连接到RNA多聚腺苷酸化(poly (a))尾巴上,随后是一个可供选择的逆转录步骤,以提高测序产量。第二步就是添加引物接头,这个接头上带有驱动测序的马达蛋白。然后,文库制备好后用于MinION测序,测序方向从RNA的3ʹPoly (A)尾到5ʹ帽子方向进行。 最初的研究表明,dRNA-seq的测序读长大概在1000bp左右,最大测序长度过超过10kb。与短读长测序相比,长读长测序的优点在于可以提高对异构体的检测,还可以用于poly(A)尾巴长度的评估,这对于可变poly(A)分析来说非常重要。Nanopolish-polya工具可以使用纳米孔测序得到的数据进行poly(A)尾巴长度的计算,除此之外还可以进行基因间以及转录异构体之间分析。分析表明滞留内含子的转录本与完全剪接转录本比较具有稍长的poly(A)尾巴。dRNA-seq它还具有检测RNA碱基修饰的潜力,为我们研究表观转录提供巨大帮助。 虽然长读长在转录本评估方面具有一些明显的优势,但也存在一些局限性。例如测序通量低,错误率多。但是,长读长技术最大的优点是可以捕捉到更多的单一转录本,但是这需要有高质量的 RNA文库。总之,这种限制会对只依靠长读长测序实验的敏感性和专一性产生影响。 长读长测序的最大限制是通量。在 Illumina上,一次RNA-seq可以得到109-1010个较小的读取长度,而在 ONT和 PacBio上,一次RNA-seq可以得到106-107个读取长度。总体来说,这些局限影响了那些完全依赖于长读长测序实验的灵敏性与特异性。长读长测序方法的主要局限就是当前的通量。这样的低通量,使长读长测序用于实验的范围受到了制约,同时也影响了基因表达水平差异检测的敏感性。但是,不是每一项实验都要求进行高深度的测序,对于那些致力于寻找异构体的学者而言,测序的长度重要性远大于测序深度。当长读长RNA-seq读长数目增加,其敏感性就会提高到与 Illumina相似的高度,并且在同一时间内也表现出较高的特异性。在保持转录本定量的同时,将 Illumina的短读长RNA-Seq和 PacBio的长读长Iso-Seq相结合,能够提高对全长RefSeq注释异构体检测的数量、敏感性和专一性。 长读长测序的另一个局限是误差较大,与已有的 Illumina平台相比,其误差率要高出一两个量级。在长读测序中产生的数据中也包括了很多插入、删除误差。尽管这种误差与识别变化有关,但是RNA-seq中的各个碱基是否被准确地识别并非那么重要,因为其目的是为了明确转录本和异构体。 PacBio SMRT测序中的随机误差可以用 CCS方法来提高测序深度来规避,即将 cDNA的长度进行选取并且和接头进行环化,将每一个分子进行多重测序。在进行了数据分析之后,将这些长读长作为单一的 cDNA子读长,从而生成一个连续的子读长序列。分子测序次数越多,其误差率越小;CCS技术可以把误差率降到与短读长相似水平。然而,如果将这种测序功能多次阅读相同的分子,那么它的通量问题就会更加严重,因为能够阅读到的惟一转录本数量越来越小。另外还有一些其它的原因制约了长读长RNA-seq方法的灵敏性。 首先,他们依靠长RNA分子来进行测序,但由于 RNA在样本制备期间会被降解或者剪切,所以不可能完美实现。这个现象在短读长RNA-seq 中也同样存在,只不过在短读长RNA-seq中是可控的,对于长读长RNA-seq而言即便 RNA分解程度很小也会对RNA-seq结果造成影响。所以,对将要进行长读长测序的研究人员而言,必须认真做好 RNA样本质控。其次,在样本制备过程中,由于技术上的问题和偏差,中位数的读长长度将会进一步受限,比如一些 cDNA的合成被阻断或者一些 cDNA是通过降解 后的mRNA逆转录而来,近些年由于一些高效逆转录酶的研发,在一定程度上改善这一问题。第三,长读长测序平台本身存在的偏差(比如,在测序芯片上的长文库会出现低散射)会减少覆盖率。长读长测序(利用 cDNA或dRNA-seq)可以生成从Poly(A)尾部到5ʹcap的跨异构体的全长转录本读长,无需进行转录本组装,因此克服了在短读长RNA-seq中对异构体检测困难的问题。采用全长 cDNA测序或dRNA-seq进行高质量的DEG分析,需要PacBio和 ONT技术平台具有更高的通量。将长读长RNA-Seq测序与高深度的短读长 RNA seq数据相结合,以进行更完整的分析。由于检测手段的不断完善和测序通量的不断提高,基因的差异性检测将会是一种常用的检测手段。长读长RNA-seq或者其他技术会给该行业带来怎样的冲击,我们拭目以待。但就当前而言, Illumina的短读长度RNA-seq仍然是主流。 下期 ,我们将聚焦的转录组的应用,敬请期待~ |