NEWS
新闻资讯
|
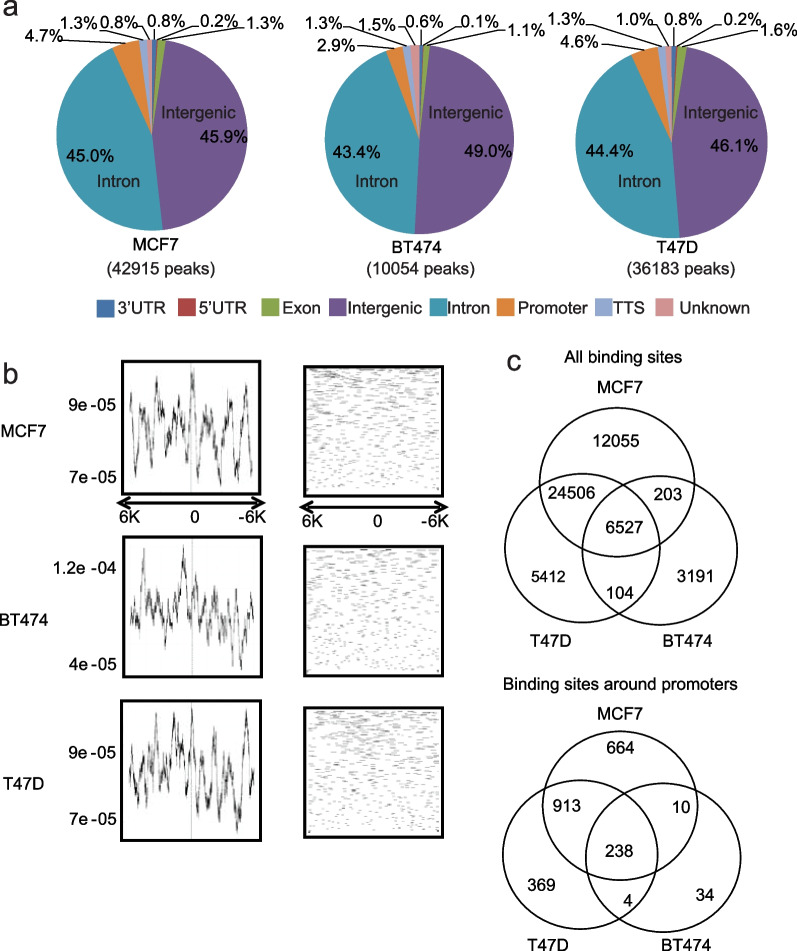
Cell Commun Signal|ChIP-seq和Bru-seq揭示转录因子GRHL2在管腔型乳腺癌中的调控网络 看看AI给你的标题打几分吧~GRHL2是粒状头样(GRHL)3个家族成员之一,其参与调控胚胎发育、细胞骨架调节、上皮细胞分化、表皮屏障形成以及表皮损伤修复等一系列重要生命过程。一些基因已经被确定为GRHL2的直接靶点,例如E-钙黏蛋白(E-cadherin)和Claudin 4。通过查看之前的ChIP-seq研究数据,发现 (1)这些不同组织之间关于GRHL2结合峰和候选靶基因一致的并不多,这表明GRHL2的调控具有组织特异性;(2)对于许多GRHL2靶基因的调控是并不是直接的,调控过程可能涉及到其他转录因子或表观遗传修饰因子。 乳腺癌是一种异质性疾病,管腔亚型占乳腺癌病例的大多数,可以通过靶向雌激素受体α (ER⍺)信号的治疗。研究表明GRHL2可能与FOXA1一样,作为一个先锋因子,促进染色质开放。GRHL2已被发现与FOXA1、GATA3和ER⍺共同占据增强子元件,以调节激素受体阳性乳腺癌中的ER⍺信号产生,但GRHL2在管腔型乳腺癌中的调控机制还有待研究。  来自荷兰莱顿大学莱顿药物研究学术中心的Zi Wang 利用ChIP-seq和Bru-seq探究转录因子GRHL2在3种管腔乳腺癌细胞系中的调控网络。 首先作者利用ChIP-seq获得GRHL2在人管腔乳腺癌细胞系MCF7、T47D和BT474中的结合位点。ChIP-qpcr作为ChIP-seq质控,验证了三种细胞系中已知为GRHL2的靶基因CLDN4。ChIP-seq结果显示GRHL2结合位点主要位于基因间区和内含子,其中~3-5%的peak位于−1000~ + 100 bp的启动子区;且Read count显示在TSS附近是没有富集的。三种细胞系共有的GRHL2结合位点有6527个,其中直接结合启动子区域的是238个(Fig.1)。
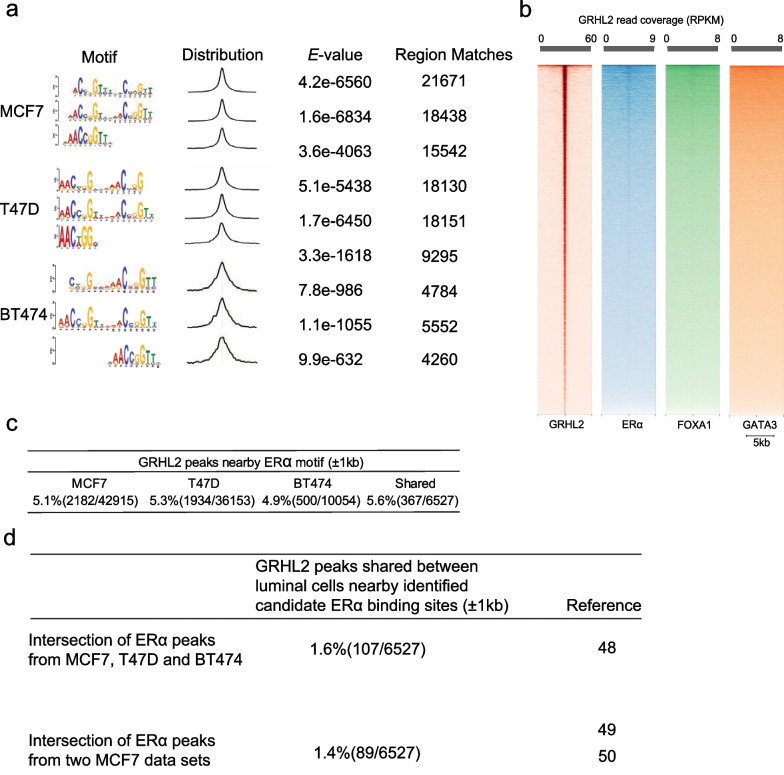
在三种细胞系中GRHL2的motif与ER⍺结合MEME-ChIP中的结果相对吻合;基于已发表的GRHL2与ER⍺、FOXA1和GATA3在靶基因motif的比较,热图可视化显示GRHL2峰值集中在GRHL2的motif[AACCGGTT]处 (图2b)。GRHL2峰仅显示ER⍺[AGGTCAnnnTGACCT]的微弱相关,FOXA1基序[TGTTT(a /G)C]的相关性更弱,并未观察到与gata3的motif相关。 由于和之前研究有所差异,针对GRHL2和ER⍺结合的共定位问题,作者对MCF7、BT474和T47D中保守GRHL2的peak±1000bp侧边区域进行了探究,以及MCF7、BT474和T47D中ER⍺的ChIP-seq数据进行交叉联合分析。在此次的研究中发现的保守GRHL2的peak中,只有一小部分(约1.5%)在之前研究中发现的腔内乳腺癌细胞中存在ER⍺结合位点。总之,这些数据表明腔内乳腺癌细胞中的大多数GRHL2结合位点与ER⍺介导的转录复合体无关。
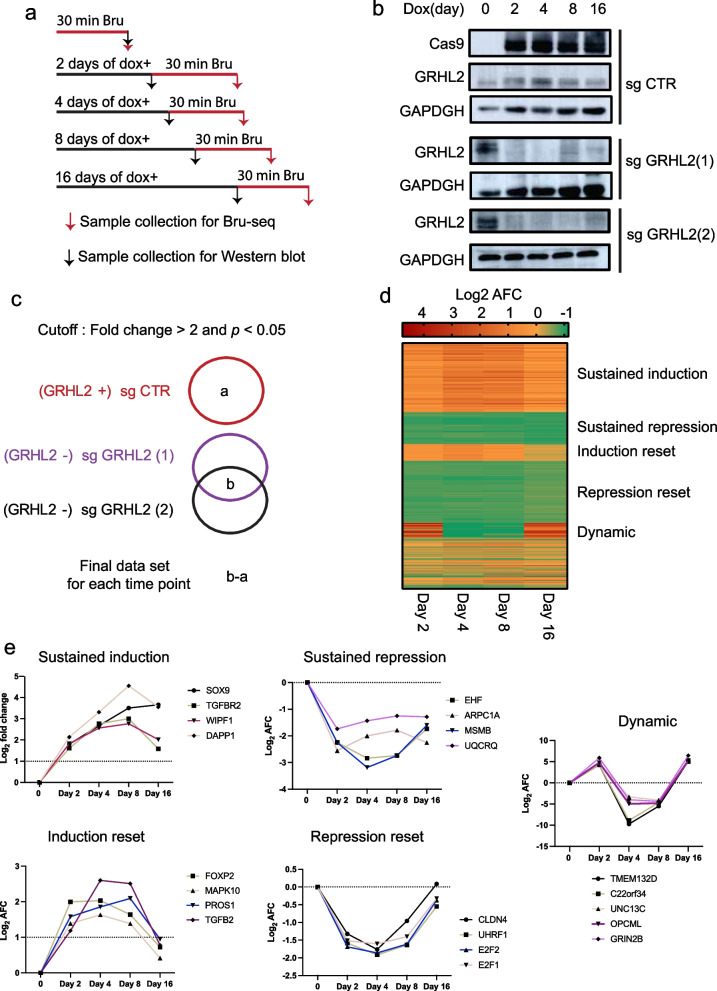
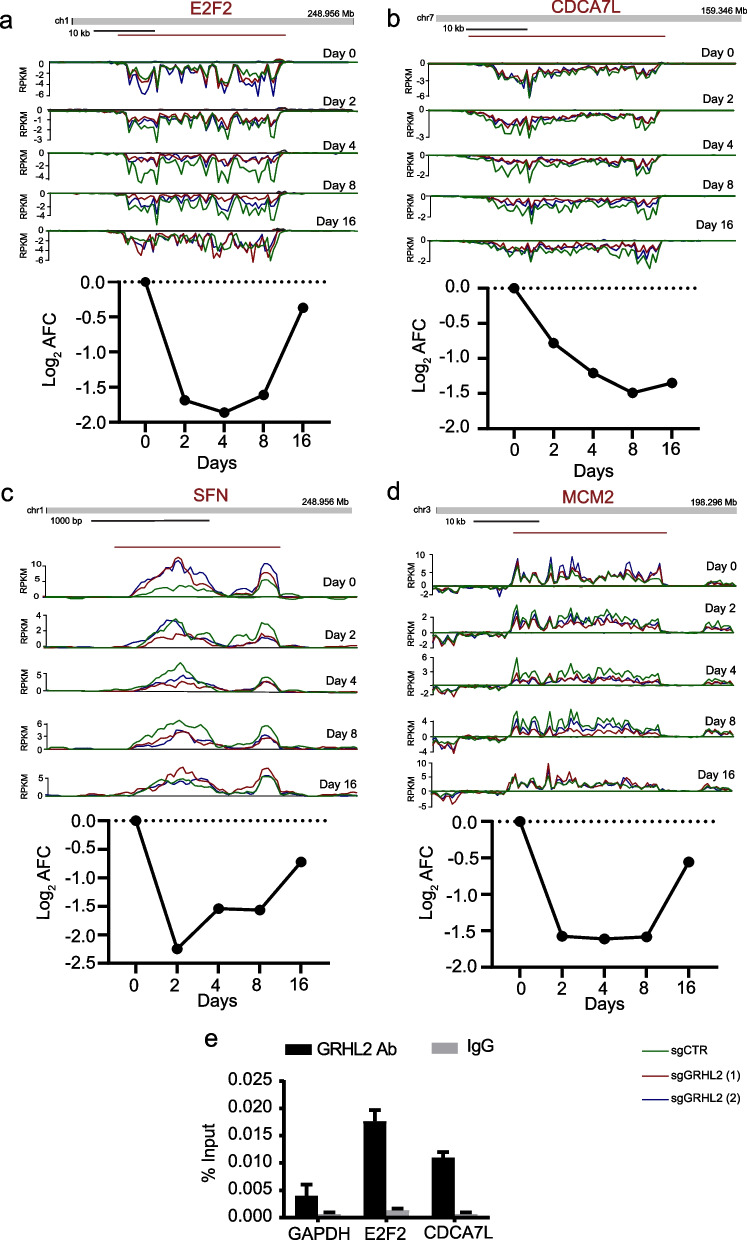
接下来,作者利用新生RNA Bru-seq研究GRHL2缺失引发的DNA转录的动态变化。作者在MCF7细胞中利用Cas9将GRHL2敲除,分别为sgCTR(对照)、sgGRHL2(1)和sgGRHL2(2))(敲除)。在GRHL2敲除后的0、2、4、8或16天,用溴吡呤(BrU)孵育细胞30分钟,以标记新生RNA,并通过Western blot并行分析Cas9的诱导和GRHL2的缺失进行检测。 为了鉴定GRHL2调控的基因,在每个时间点,测定了在两个sgGRHL2和sgCTR样本中测定基因表达情况。在两个sgGRHL2样本中,GRHL2缺失后至少一个时间点有262个基因上调,226个基因下调,而sgCTR样本中则没有变化。将相关基因表达按照表达模式差异分成5个cluster,GRHL2结合位点的基因子集没有表现出一定的偏好。其中TGFß通路相关基因TGFB1, TGFB2和TGFBR2随着时间延长RNA合成持续上调或随后恢复到基线。参与细胞周期进程的E2F转录因子及编码上皮紧密连接蛋白的CLDN4基因在GRHL2缺失后RNA合成持续下调,随后恢复到基线水平。
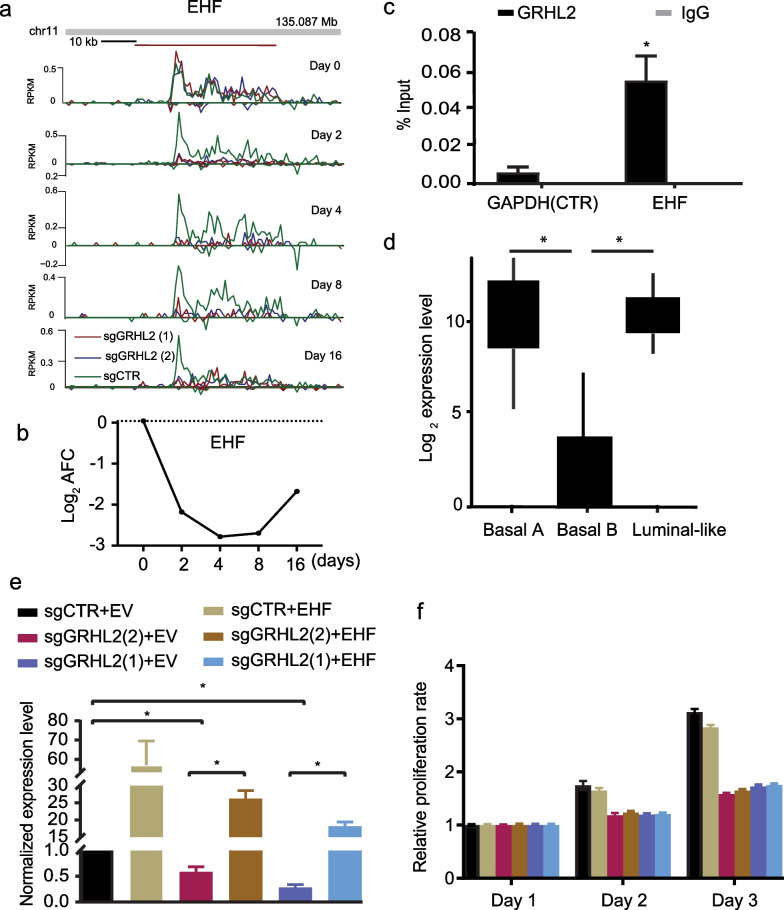
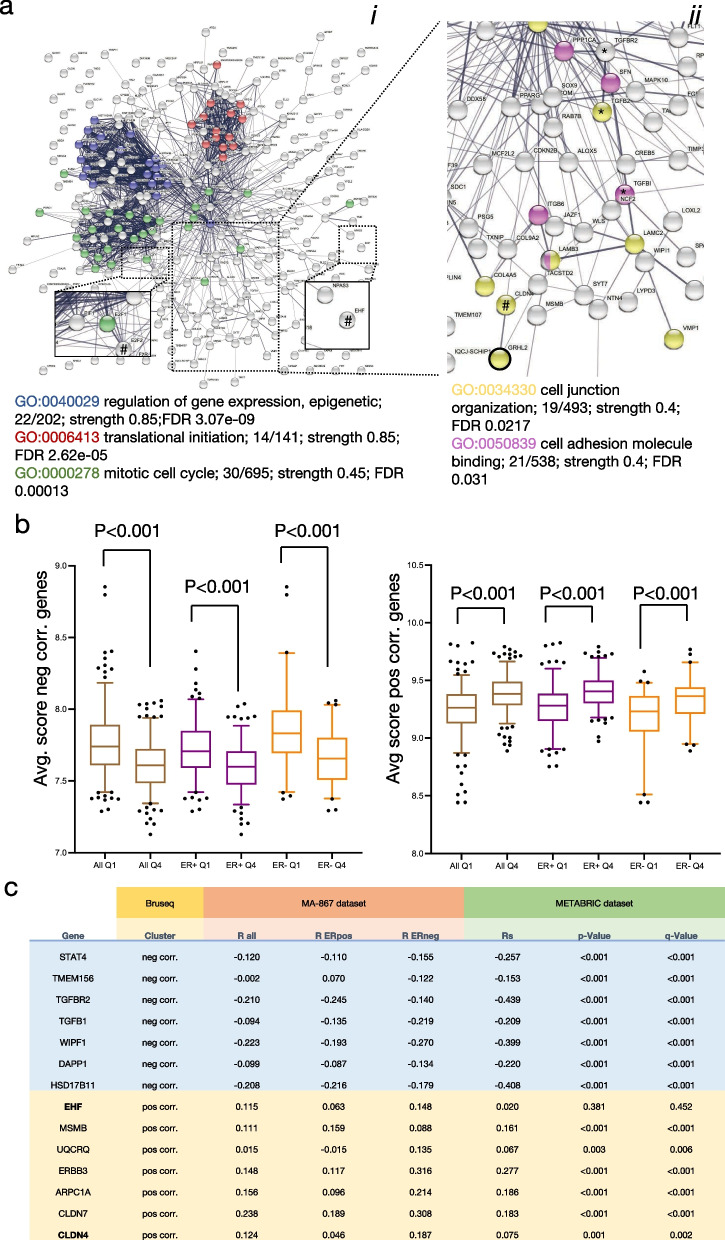
通过MCF7细胞ChIP-seq中peak在启动子区和Bru-seq联合分析鉴定得到53个基因,进一步与三种乳腺癌细胞ChIP-seq共有基因取交集得到最后9个基因。结果表明GRHL2缺失表现出转录应答的基因是通过与增强子元件的直接相互作用或间接调控的。 研究数据表明,乳腺癌中EHF作为GRHL2的直接靶基因,与GRHL2呈负相关。ChIP-qpcr验证GRHL2与EHF基因启动子区域结合;Bru-seq结果显示GRHL2缺失后EHF转录迅速且持续降低。查阅前人发表的52个人类乳腺癌细胞系的RNA-seq数据显示,在基础B亚型中EHF 的RNA水平是下降的。GRHL2缺失导致MCF7细胞生长迅速减少,作者在GRHL2缺失细胞中过表达EHF,发现并不能挽救GRHL2 KO MCF7细胞的细胞增殖。EHF、E2F2和CDCA7L是GRHL2在基因启动子处直接转录调控的候选靶点基因,在Bru-seq中显示在GRHL2缺失后被迅速抑制。
Fig.4 EHF是GRHL2直接调控的基因
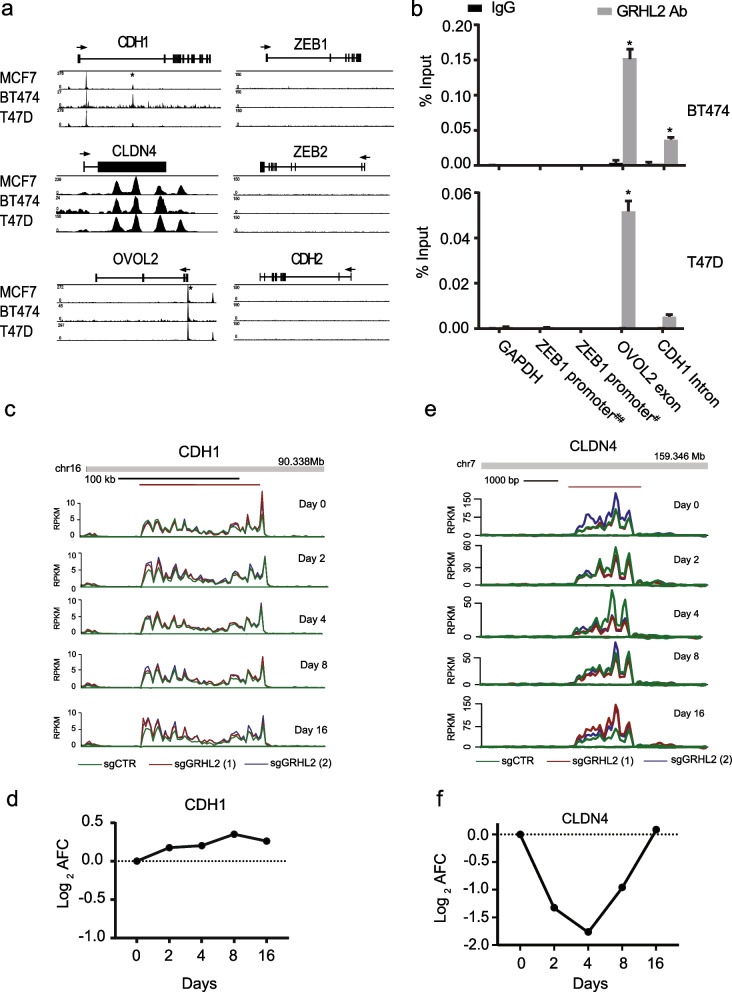
对于GRHL2靶标EMT相关基因,CLDN4以及 E-cadherin (CDH1),ChIP-seq结果中显示GRHL2结合在CDH1内含子区,通过ChIP-qPCR验证了这一结果。尽管在早期的报道中发现GRHL2可以转录激活CDH1,但在GRHL2丢失后的前16天内,并未出现CDH1新生RNA合成的下调(Fig. 6c, d)。而GRHL2的结合在CLDN4和OVOL2的启动子区域是保守的(图6a, b;)。CLDN4在编码区和非编码区也显示出多个GRHL2结合位点。在GRHL2耗尽后的2,4,8天,CLDN4转录被抑制,但在16天恢复(Fig. 6c, d),而OVOL2不受影响。在ZEB1或ZEB2的启动子或其他区域也没有GRHL2结合peak。这些结果表明CLDN4是GRHL2的直接靶基因,而CDH1、ZEB1或ZEB2不太可能是管腔乳腺癌细胞中GRHL2的直接靶基因。
Fig.6 GRHL2对EMT相关基因的调控 作者使用了867名未经治疗的乳腺癌患者(MA-867)数据,并根据GRHL2表达水平分析TGFß信号通路相关基因,得到与本次研究一致的相关性。
总 结 |