NEWS
新闻资讯
|
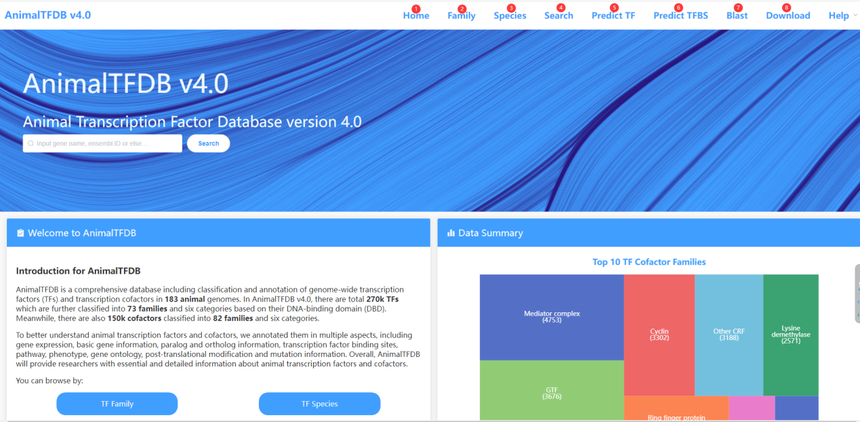
AnimalTFDB v4.0 手把手教您使用动物转录因子数据库转录因子(Transcription factors,TFs)在控制基因表达中起着关键的作用。对TFs的系统性识别和注释,随后是TF数据库的构建,可能充当了研究转录因子的功能和进化的有用资源。动物转录因子数据库AnimalTFDB4.0(http://bioinfo.life.hust.edu.cn/AnimalTFDB4/#/)对97个动物基因组的转录因子(Transcription Factor)和转录辅助因子(Transcription cofactor)进行了归纳整理。基于DNA结合结构域,将动物转录因子分成了73个基因家族,将转录辅助因子分成了83个基因家族。此外,动物转录因子分为六大类(Basic Domain Group、Zinc-Coordinating Group、Beta-Scaffold Factors、Helix-turn-helix、Other Alpha-Helix Group和Unclassified Structure),动物转录辅助因子也分为六大类(Co-activator/repressors、Chromatin Remodeling Factors、General Cofactors、Histone-modifying Enzymes、Cell Cycle和Other Cofactors)。 1. 数据库主页布局介绍
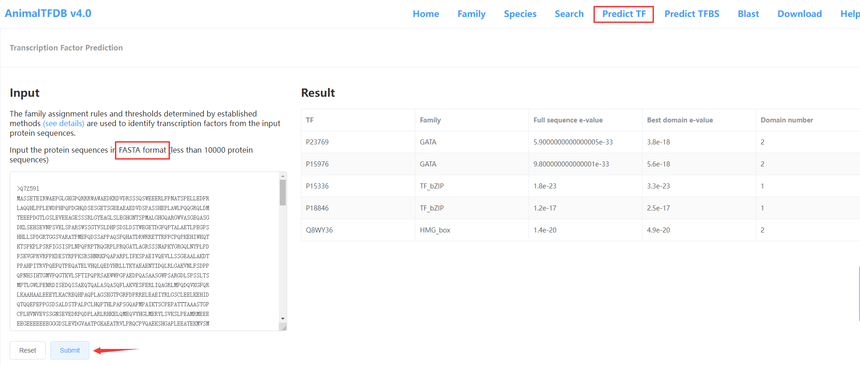
① 点击Home即可跳转到主页面。 ② 点击Family跳转到TF分类家族页面。展示TF家族分类信息。 ③ 点击Species跳转到根据物种分类页面。展示根据不同物种归类的TF信息。 ④ 点击Search跳转到搜索页面。输入基因ID信息查询相关注释信息。 ⑤ 点击Predict TF跳转到TF预测页面。输入fasta格式的蛋白序列文件,基本比对方法预测TF。 ⑥ 点击Predict TFBS跳转到TF结合位点预测页面。输入fasta格式的核苷酸序列文件,基本比对方法预测TFBS。 ⑦ 点击Blast跳转到比对搜索页面。输入fasta格式的核苷酸或蛋白序列文件,指定物种进行比对。 ⑧ 点击Download跳转数据下载页面,可以下载各个物种分类的TF相关蛋白序列文件。 2. 查询转录因子案例 2.1 已知序列点击Predict TF跳转到转录因子预测页面,在文本框中输入fasta格式(fasta 格式:大于号>后紧跟序列名,换行后是序列)的蛋白序列信息(只支持蛋白序列文件),再点击Submit按钮即可将任务提交给后台运行,如果序列数据不多很快就可以反馈结果。
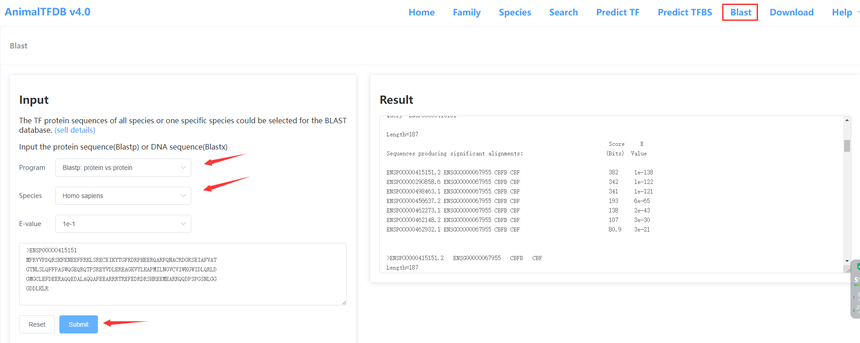
如果只有核苷酸序列文件,同样可以比对预测。点击Blast进入比对页面,在program选项中选择Blastx模式,输入fasta格式的核苷酸序列文件,选择物种信息后点击Submit按钮即可将任务提交给后台运行。随后返回比对结果。
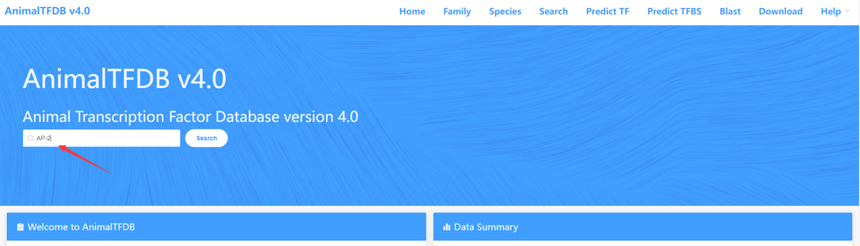
2.2 已知转录因子名字已知某个转录因子名字,以AP-2为例。在主页的右上角有一个搜索框,输入AP-2关键词后点击搜索按钮,即可得到AP-2相关的页面结果信息,结果页面展示了AP-2相关基因的ID、物种和注释信息。可以通过物种筛选按钮,根据物种类别进行筛选。 还有另外一个方法,点击进入Family主页面,页面展示的是不同分类的家族TF信息。找到AP-2家族后点击进入家族页面,页面展示了不同物种的AP-2转录因子信息,点击某个物种即可查询该物种TF信息。
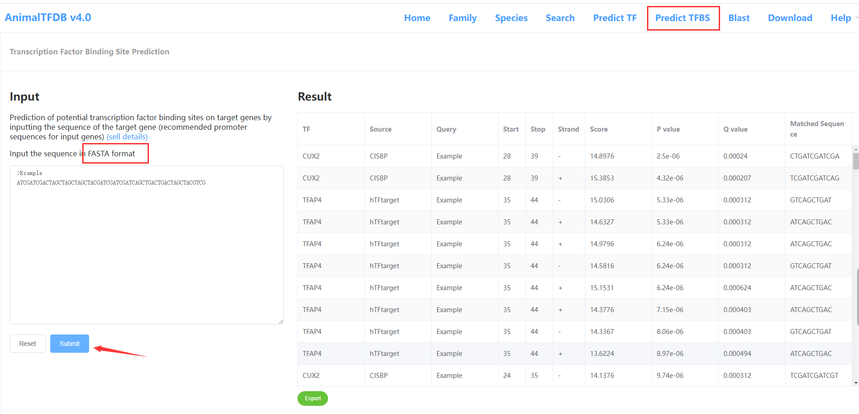
3. 转录因子结合位点预测 已知一段DNA序列查询其潜在的TF binding site,点击Predict TFBS进入主页面,输入fasta格式的核苷酸序列,点击Submit按钮即可将任务提交给后台运行,结果反馈预测的TF binding site信息。还可以根据TF家族分类进行筛选。
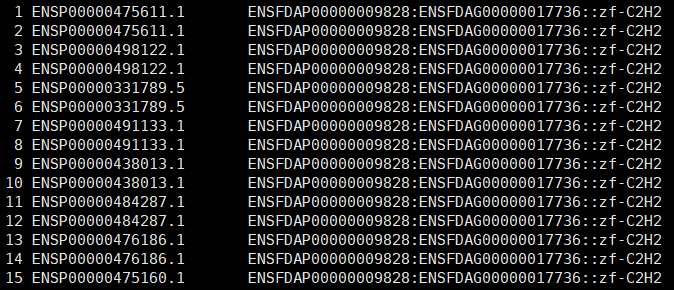
4. 用linux服务器进行批量分析 4.1转录因子鉴定在AnimalTFDB线上数据框下载所有动物转录因子蛋白序列文件,基于blast软件进行比对分析,得到同源序列比对结果,则预测推断这些序列具有同样的TF功能。比对命令如下: blastp –query query.pep.fa -db /path/animal_TF -out blast_out.xls -outfmt 6 -num_threads 10 -evalue 1e-10 比对完成后可得到如下文件,第二列为预测的TF结果。
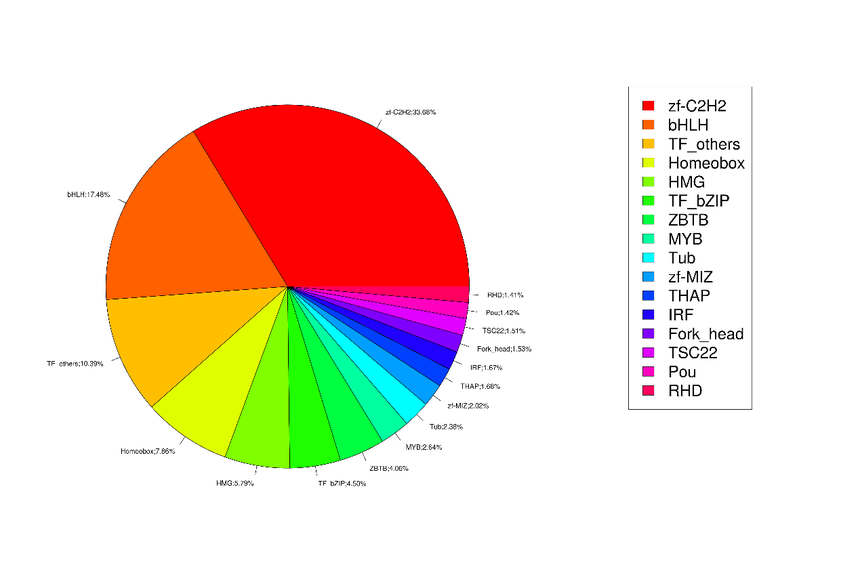
还可以对转录因子家族进行统计并简单作图如下:
在得到目标的转录因子之后,可以根据转录因子motif网站 http://jaspar.genereg.net/ 提供的motif序列,利用homer[2]软件对目标序列(peak或者Promoter区域的序列)进行motif分析,得到目标区域的已知转录因子motif。然后根据peak或promoter关联的基因及其注释筛选出motif结合基因,对这些基因用clusterprofiler 软件做GO富集分析,最后利用Cytoscape做成网络互作图。示例结果如下:
|