NEWS
新闻资讯
|
Cell | 利用SiTomics串联表观修饰,蛋白组和基因组题 目:Linking chromatin acylation mark-defined proteome and genome in living cells 发表日期:2023年3月2日 发表期刊:Cell 影响因子:66.85 2023年3月2日北京大学化学与分子工程学院北京分子科学国家实验室合成与功能生物分子中心陈鹏团队在期刊Cell(IF: 66.85)发表题为“Linking chromatin acylation mark-defined proteome and genome in living cells”的研究论文。作者开发了一种单位点解析的多组学(SiTomics)策略,用于系统地映射动态修饰,随后对由特定染色质酰基定义的染色质化蛋白质组和基因组进行分析。 对未受干扰的染色质上的组蛋白修饰进行原位分析是一种可广泛应用且具有可编程位点特异性的策略,这一直是非常理想但具有挑战性的。在此,作者开发了一种单位点解析的多组学(SiTomics)策略,用于系统地映射动态修饰,随后对由特定染色质酰化定义的染色质化蛋白质组和基因组进行分析。通过利用遗传密码扩展策略,SiTomics工具包揭示了短链脂肪酸刺激后的明显丙酰化(例如H3K56cr)和β-羟基丁酰化(例如H3K56bhb),并建立了染色质酰化标记定义的蛋白质组、基因组和功能之间的联系。作者发现GLYR1可以作为一个独特的相作蛋白调节H3K56cr的基因定位,以及在bhb介导的染色质调控下发现升高的超增强子库。SiTomics提供了一种平台技术,用于阐明“代谢物-修饰-调控”轴,可广泛应用于超越酰化以及组蛋白以外的蛋白质的多组学分析和功能分析。 作者首先使用SiTomics的位点分析模块来量化HEK293T细胞中组蛋白酰化位点和在SCFA应激下的动态变化。通过添加短链脂肪酸(SCFA)前体物质(如:巴豆酸钠[Nacr]、β-羟基丁酸钠[Nabhb])并达到合适的浓度,促进乙酰辅酶A的水平升高,从而增强组蛋白的乙酰化。对酸提取的同位素标记的组蛋白进行基于溶液或凝胶的蛋白质组学,用胰蛋白酶或GluC消化进行定量MS分析(图1B),结果显示出良好的敏感性和覆盖率,并对已知修饰位点进行了验证。此外,MS分析还揭示了四个以前未报道的修饰位点K36、K37、K64和K116(图1C)。所有已鉴定的肽段揭示了组蛋白H3蛋白近端修饰之间的潜在串扰。揭示了组蛋白H3蛋白质相邻修饰之间的潜在相互作用。尽管赖氨酸巴豆酰化(Kcr)和β-羟基丁酸化(Kbhb)修饰显示出不同的分布模式,但这两种修饰在H3K56上都显著增加(图1D和1E)。 值得注意的是,由于代谢产物诱导的动态组蛋白修饰通常与染色质可及性相关,以调节基因表达。H3K56位于核小体的出入口位点,是控制核小体拓扑结构(如DNA呼吸)的关键位置,与转录调控有关。鉴于对该位点修饰的功能研究仍然有限,作者在含有修饰和未修饰的H3K56进行了体外重建完整核小体,并对核小体进行可及性分析(即Pst1消化),进一步说明了它们不同的DNA解旋效应(图1F)。  图1. SiTomics策略概述和处理后代谢产物特异性组蛋白酰化动态变化的比较分析。 为了开发SiTomics的site-link和site-seq模块,作者首先合成了Kacyl和K*acyl(位点特异性结合的光亲和酰基赖氨酸)探针(图2A),其中包括一系列组蛋白赖氨酸乙酰化(Kac)、丙酰化(Kpr)、丁酰化(Kbu)、巴豆酰化(Kcr)、2-羟基异丁酰化(Khib)和β-羟基丁酸化(Kbhb)。作者发现N-Boc-photolysine与hib-琥珀酰酯反应良好,工作步骤更简单。同时,为了进一步确保K*acyl探针的特异性和生理相关性,作者还开发了基因编码的、化学保护的光亲和赖氨酸(PABK*)(图2A),允许在经过TCO介导的化学解除后,在特定位点引入“未修饰”的光亲和赖氨酸(K*)。 然后,作者对包含211个不同活性位点突变组合的PylRS突变文库进行了基于GFP报告系统的筛选,以寻找能够识别这些Kacyl/K*acyl类似物的PylRS变体(图2C)。这些探针在大肠杆菌和哺乳动物细胞中(图2F)均可以被识别。作者筛选的PylRS变体在Kpr、Kbu或Khib的琥珀酰突变抑制效率方面均比之前报道的变体具有更高的效率。细胞定位分析、分离鉴定和MS/MS分析进一步证实它们成功地整合到细胞内的染色质中,并在特定位点形成解除的K*(图2G-2J)。同时,利用重标氨基酸探针证明了修饰基团在细胞中的稳定存在时间。这些详实的数据确证了带有定点翻译后修饰的组蛋白能够在活细胞内稳定存在并被有效整合入染色体当中。 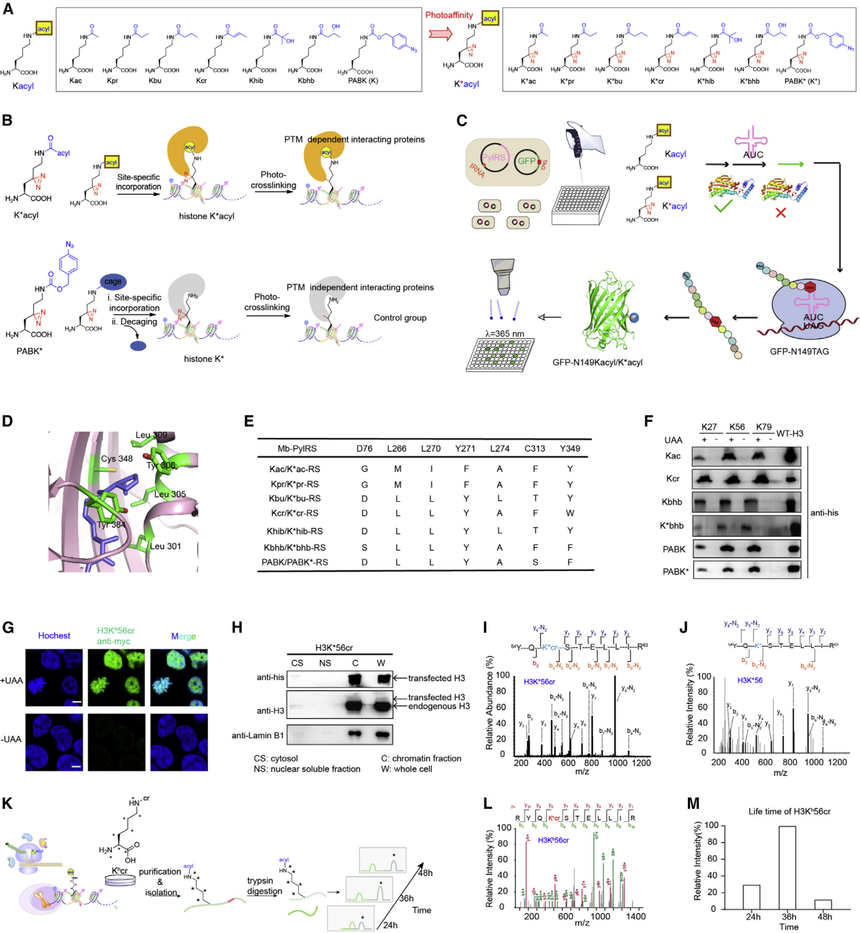 图2. site-seq和 site-link工具包的开发 为了验证site-link工具包的PTM和位点依赖性,作者采用了定量和比较蛋白质组学策略,并在具有高再现性的生理染色质的背景下获得了每个携带PTM的组蛋白变体的特异性相互作用组分。首先对H3K*56ac和未修饰的H3K*56进行组间比较分析,在此过程中,在H3K*55ac位点连接中鉴定出组蛋白乙酰化-BRD4(含溴代烷的蛋白4)的已知reader蛋白,而H3K*66仅与少数蛋白交联,如组蛋白和伴侣蛋白(图3A、3G)。此外,发现H3K*56ac与CHAMP1交联,CHAMP1是有丝分裂中染色体分离的调节因子,以及转录激活剂的排列,如CREB5、ONECUT2、POU3F2和SF1,与已报到的基因激活作用一致(图3A、3G)。 作者接下来研究了不同组蛋白位点上的Kcr特异性相互反应组(图3B–3D),在新鉴定的修饰位点上揭示了一系列结合伴侣(图3B),这与巴豆酰化在精子形成中的功能一致,包括FOXJ3,以及先前报道的H3K27cr 的reader蛋白YEATS2。但在该位点未鉴定出已知的组蛋白修饰writer或eraser(图3B、3C、3G)。H3K*79cr上的光交联也是如此,H3K*79 cr是H3球状结构域中的一个埋藏位点,参与结合组蛋白H4(图3D、3G)。相反,作为一个更具动态性的修饰位点,对H3K*56cr的位点连接研究揭示了更多的转录调节因子,包括MBB1A和ONCE2,以及几种writer/reader/eraser,包括ANM1、ANM5、GLYR1、SPIN1、HDAC1和HAT,反映了其内在的动态特征和积极参与转录激活(图3C、3D和3G)。 为了进一步证明site-link即使在同一位点也能区分不同酰化部分的相互作用组,作者在H3K*56bhb上进行了光交联实验,并将收集的蛋白质组学数据集与上述H3K*56cr数据集进行比较分析。与H3K56ac类似,MS/MS分析显示共有28种重叠蛋白主要参与组蛋白组装或转录激活,如INO80、SPT16、GATA6和TIF1B,这反映了这些组蛋白酰化和乙酰化之间的某些共性。同时,H3K56cr和H3K56bhb的互作蛋白有很大不同,H3K56cr(83种蛋白质)和H3K56bhb(115种蛋白质)分别有一个独特的互作蛋白库(图3E、3F)。进一步分析表明,由于CCNB1、CCNB2、CDK2和SND1是互作蛋白,H3K56bhb可能在细胞周期调节中发挥潜在作用(图3G)。  图3.利用定量和比较蛋白质组学验证site-link工具包 根据蛋白组数据,作者利用H3K56cr作为示例用来验证SiTomics是否可以利用组蛋白修饰建立蛋白组和基因组之间的联系。首先,作者在H3K56cr的互作组列表中挑选SSRP1(结构特异性识别蛋白-1)作为示例蛋白,它是高度保守的二聚体FACT(促进染色质转录)复合物的一个亚基,在各种染色质相关过程中起着关键作用。作者证实H3K*56cr核提取物与SSRP1以及重构的FACT复合物在紫外线照射下交联(图4A)。同时,肽结合实验表明,FACT复合物与H3K56cr和未修饰的H3K56具有类似的结合力(图4A)。 接下来,作者发现一系列已知与H3K4me3相互作用的H3K56cr结合蛋白,例如SPIN1(H3K4me3的reader),YY1(与H3K4me3修饰启动子相关的增强子-启动子环的结构调节因子),PAXI1(组成组蛋白甲基转移酶复合物所需的组件H3K4me3),RBBP5(促进MLL蛋白甲基转移酶活性的保守因子)和MEN1(通过染色质环路与H3K4me3相关的因子)。由于H3K4me3是一个主要位于基因启动子区的基因激活标记,所以ChIP-qPCR被用来检测H3K56cr修饰的基因组定位。作者使用H3K27ac作为对照,并且预修饰的H3K27ac确实在增强子区域富集。通过使用携带PiggyBac转座子介导的PylRS整合系统(STAR方法)的相同稳定细胞系,对H3K56cr的ChIP-qPCR分析显示在启动子区域富集,这与pan-Kcr的启动子定位一致。为了进行进一步的功能表征,用SPIN1孵育重建的不对称核小体,同时包含H3K56cr和H3K4me3的核小体(图4B,lane 10)比仅包含H3K56cr或H3K4me3(lane 5和lane 8)的核小体DNA更容易获得,表明它们具有协同效应。 最后,作者还观察到Kcr的很大一部分峰值位于基因体区域,这与H3K56ac有所不同。因此,作者进一步研究了位于基因体区域的H3K56cr互作蛋白,如GLYR1。实验表明GLYR1选择性地与H3K56cr相互作用,但对其他H3K56酰化作用较少(图4C)。通过表面等离子体共振(SPR)分析和随后与H3K36me3的比较研究,还验证了特异性结合亲和力。使用pLink搜索体内交联肽段,确定了GLYR1上AT钩结构域内的R174作为它们的接合部位(图4E)。GLYR1的ChIP-seq分析显示基因体区域的峰值占很高的百分比(73%)(图4F)。利用光亲和探针的光激活特性,作者在不同时间点进行了时间分辨光交联实验,使用等量的染色质化H3K56cr(如图4G所示)。有趣的是,尽管H3K56cr在Kcr探针结合后24小时及以后能有效地整合到核小体中(图2M),但H3K56cr与GLYR1之间的交联直到36小时及以后才变得明显(图4G),表明GLYR1的参与可能发生在H3K56cr整合到染色质之后。 因此,作者分别在启动子和基因体区域验证了H3K56cr与SPIN1和GLYR1的直接和特异性功能和相互作用。根据GeneCards、PubMed和UniProt数据库中报告的功能和基因组定位,进一步总结了从H3K*56cr 的site-link结果中获得的染色质结合候选蛋白(图4H)。  图4. SiTomics策略在活细胞中将染色质标记依赖的蛋白质组和基因组关联在H3K56cr上的展示 作者通过对H3K56bhb进行site-seq分析以获得关于SiTomics整合组蛋白修饰的更多功能见解。研究发现,H3K56bhb在基因组区域的富集程度高于未修饰的H3K56。H3K56bhb的染色质占位信号与基因表达弱相关,与H3K27ac(基因激活标记)相比,但与H3K9me3(抑制标记)无关。基因集富集分析(GSEA)显示,H3K56bhb位点测序峰值在基因组中的分布不均匀,信号较强的峰值在mRNA处理和信号通路(如细胞因子产生)以及参与免疫应答的细胞激活等途径中富集得更好。研究还通过ATAC-seq分析发现,H3K56bhb在染色质区域的沉积可能与其本身的特异性有关。结果表明,与携带未修饰H3K56的细胞相比,携带H3K56bhb的细胞在H3K56bhb峰值位点的染色质可及性显著增加,表明整合的H3K56bhb具有功能。此外,这些增加的位点与H3K56bhb整合位点的重叠比率高于随机选择的区域。在排除核小体占位效应的干扰后,研究发现H3K56bhb在ATAC-seq增加的峰值处的位点测序密度显著高于不变峰值。 作者对表达H3K56bhb或未修饰H3K56的细胞进行Hi-C分析。Hi-C相互作用频率分析表明,短距离相互作用减少,而长距离相互作用增加。通过使用之前发布的流程,研究人员检查了A/B区室、TADs和loops的影响。室间分析显示A-A和B-B区室间隔略有减少,A-B和B-A区室间隔增加。此外,meta-TAD和loop分析表明,H3K56bhb的整合导致meta-TAD和APA信号略有减少。具体实例也一致地显示出loop强度略有减弱。最后,通过比较ATAC-seq增加位点和不变位点处的loop anchor相互作用的变化,研究发现H3K56bhb的整合导致这些位点的ATAC-seq峰值增加,并在loop anchor相互作用中表现出更明显的减少,暗示3D染色质组织的特异性松散。这些结果与作者的推理一致,即携带基因编码赖氨酸酰化的预修饰组蛋白可以以修饰依赖的方式沉积在染色质上,并具有修饰特异性功能,类似于相应内源修饰的位置。 最后作者使用RNA测序(RNA-seq)分析进一步阐述各种SiTomics整合组蛋白修饰的PTM特异性功能结果。作者构建了基于荧光强度筛选的PylRS表达稳定细胞系,以实现这些组蛋白变体的等量表达,这对于并行比较至关重要。值得注意的是,聚类分析表明,过表达未修饰的H3K56及其整合到染色质中对基因表达几乎没有影响,而预修饰组蛋白的引入导致基因调控模式发生变化。例如,H3K56cr上调的基因参与抗原呈递和代谢过程。进一步的比较分析显示了不同的调控模式,如H3K56ac和H3K56bhb之间的免疫应答途径(如抗原加工和呈递)、代谢过程(如脂肪酸β氧化)以及细胞周期调控中呈现出的不同模式。  图5. H3K56bhb的基因组定位及其在生理染色质上的功能的基因组测序探索与验证 β-羟基丁酸(bhb)作为关键的酮体成分在代谢生理稳态中的中心调节作用,其潜在机制尚不清楚。由于最近报道bhb在活细胞中通过形成bhb-CoA导致组蛋白Kbhb修饰的产生,作者决定关注该修饰,以检查携带特异性Kbhb修饰的组蛋白如何调节染色质和基因表达。使用全Kbhb抗体的免疫荧光成像显示,Nabhb处理引起细胞核内凝聚物的形成,而Nacr则没有这种效果。作为主要受影响的组蛋白Kbhb位点之一(图1E),H3K*56bhb的site-link显示与人类核蛋白质组相比,相对富集与相分离相关的蛋白质。基于GO分析,H3K56bhb可能在与细胞周期相关的相变中发挥作用。作者选择了CCNB1(Cyclin B1)进行验证,这是一个对G2/M有丝分裂过渡控制至关重要的蛋白质。肽结合实验验证了site-link结果的可靠性,发现CCNB1与H3K56bhb和H3K56ac相互作用,但与H3K56cr没有相互作用。接下来,作者关注在增强子区域具有高共定位倾向的内在无序区域(IDR)-含有的蛋白质,如BRD4,它可能将相分离与基因表达控制联系起来。通过肽结合实验、ITC滴定和EMSA(电泳迁移率变化分析)验证了这种相互作用。使用Rosetta进行的计算建模研究也得出了一致的结论,进一步支持H3K56bhb与BRD4之间的相互作用。 作者除了发现H3K56bhb与BRD4结合外,还发现基于site-seq分析,H3K56bhb富集在超级增强子、增强子和启动子区域。同时,H3K56bhb与共激活因子(例如BRD4和MED1)或活性增强子指示物(例如H3K27ac)的共定位也很好。同时,在含有H3K56bhb的细胞中,共激活因子的染色质结合信号显著高于未修饰的H3K56,而BRD4和MED1的总蛋白水平没有观察到变化。例如,与未修饰的H3K56整合相比,H3K56bhb整合导致BRD4、MED1和H3K27ac在TAOK1基因座的占据率增加。进一步的比较分析表明,它在超级增强子中特异性富集,但H3K9bhb没有这种富集。同时,作者还注意到,H3K56bhb上调的基因具有比未修饰的H3K56更高的Pol II ChIP-seq峰值强度和Pol II行进比率。 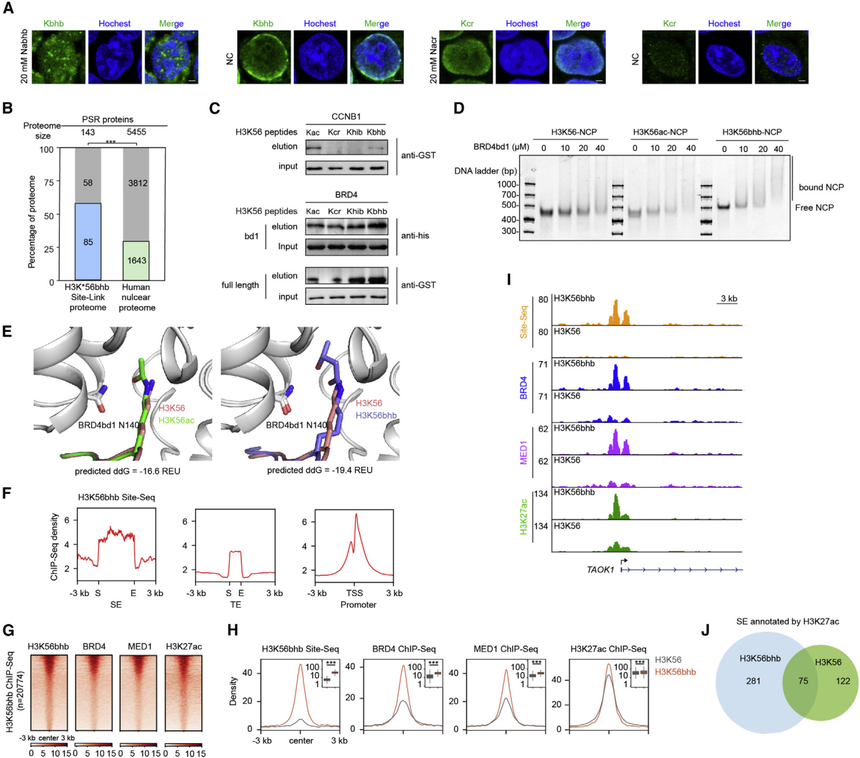 图6. SiTomics揭示了H3K56bhb与BRD4的相互作用以及在活细胞中超级增强子的富集 作者通过联合免疫沉淀实验研究了Kbhb修饰的组蛋白与超级增强子凝聚共激活因子之间的相互作用。结果发现,尽管在Nabhb处理的细胞中BRD4、MED1和H3K27ac的总蛋白水平没有改变,但与BRD4共沉淀的组蛋白H3增多。同时,BRD4和MED1与组蛋白Kbhb和H3K27ac的修饰水平呈优先增加和富集,Nabhb处理下更为显著。进一步的MS/MS分析显示,H3K56bhb是所有与BRD4共沉淀的特异性组蛋白Kbhb中最富集的标记。这些观察结果进一步表明,H3K56bhb与BRD4相关染色质之间优先连接。 ChIP-seq分析也得到了一致的结果。在Nabhb处理下,H3K27ac和共激活因子的峰值都显著增加,同时,获得的H3K27ac增强子在H3K56bhb整合位点显著富集,特别是分布在超级增强子上。同时,在超级增强子区域内,BRD4、MED1和H3K27ac的ChIP-seq信号密度显著增高。 接下来,作者试图探究哪些因子在这些位置负责转录机制的密集组装。从Cistrome数据库中下载了254个HEK293/HEK293T细胞中的已发表ChIP-seq数据,并使用回归分析确定与超级增强子变化相关的因子。结果显示,P300和BRD2的峰值位置与这些在基因组上增加的H3K27ac峰区域具有最高的相关性。P300的正相关性与其作为H3K27ac的写入酶和增强凝聚分区的作用一致,同时也与最近报道的组蛋白Kbhb潜在写入酶的作用相一致。 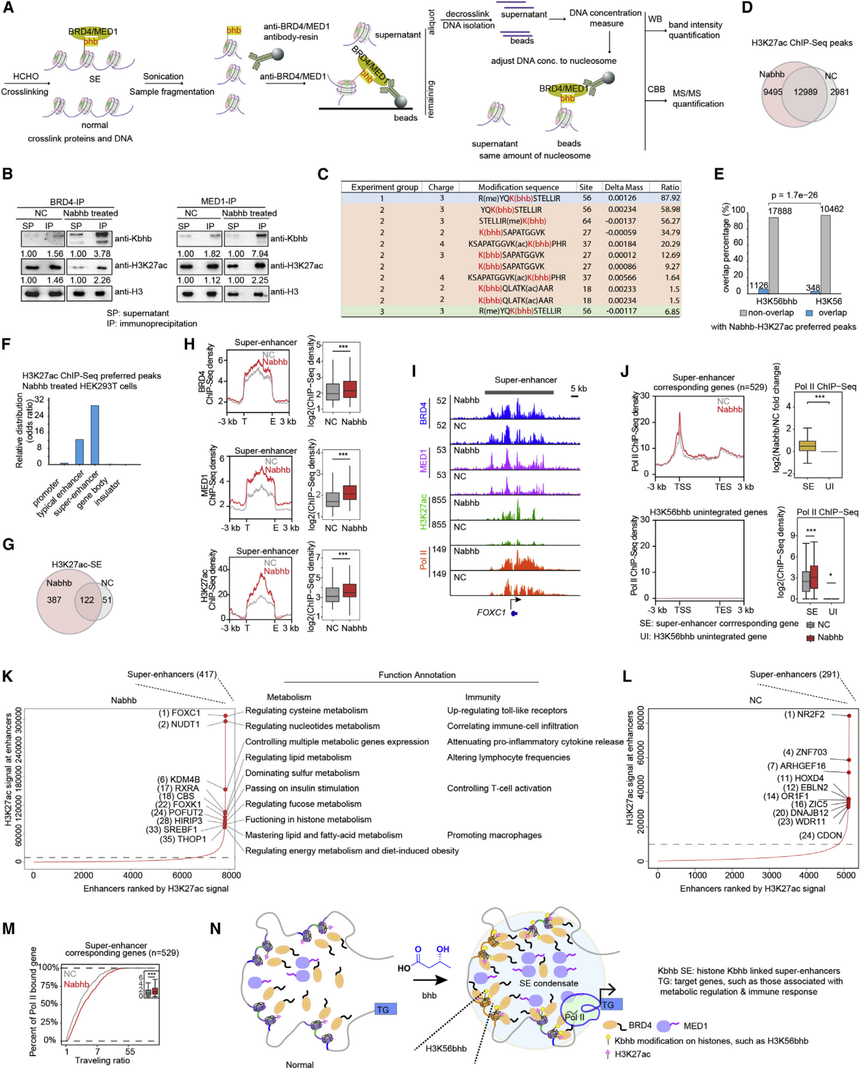 图7. 与组蛋白Kbhb连接的超级增强子有助于解释酮体β-羟基丁酸盐介导的染色质调节过程中的活性转录 为了进一步了解这些受影响的超级增强子如何在酮体bhb介导的染色质调控中发挥作用,研究人员分析了基因表达和转录装置(如Pol II)的占用情况。首先,RNA-seq表明在Nabhb处理下,mRNA表达谱优先增强。与H3K56bhb RNA-seq的相关性分析进一步证实,与Nabhb处理的细胞相比,含有H3K56bhb整合的细胞具有相似的转录趋势,涉及免疫应答中基因的上调,例如I型干扰素信号通路、外源性肽抗原的抗原处理和呈递以及中性粒细胞激活。这与酮体在免疫激活中的强大功能一致。值得注意的是,这些显著增加的基因与较高的Pol II占用相关。 接下来,将所有超级增强子分配到其关联的基因(n = 529),发现这些基因在Nabhb处理后的细胞中确实具有更大的Pol II占用部分,但对于未整合H3K56bhb的基因则不是这样。traveling ratio的显著增加再次表明,这些超级增强子可能促进活跃的转录调控。在含有H3K56bhb的细胞中观察到类似的结果。这些基因的GO分析表明,它们参与代谢调控(例如,脂肪细胞分化)以及免疫调控(例如,细胞对糖皮质激素和皮质类固醇刺激的反应以及类固醇激素)。 总之,SiTomics揭示了特异性组蛋白Kbhb(如H3K56bhb)与超级增强子区域的关联,这是酮体bhb引发的主要受影响的修饰之一,并在代谢压力下进一步揭示了染色质调控和转录调控过程中超级增强子的作用。 |












