NEWS
新闻资讯
|
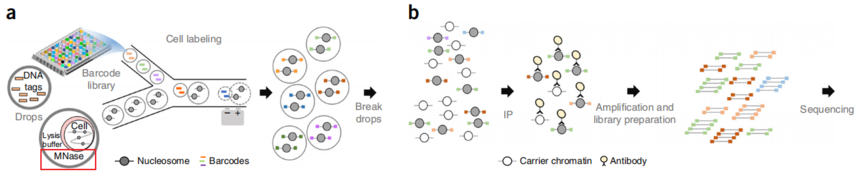
单细胞ChIP-seq,引领单细胞测序新方向染色质免疫共沉淀测序(ChIP-seq)是研究表观遗传调控的重要手段之一,它广泛用于全基因组组蛋白修饰、转录因子和其他蛋白-DNA互相作用研究。ChIP-seq作为爱基百客的王牌产品,已用多篇项目文章证明实力。 组织水平的ChIP-seq技术需要使用超声/酶切打断交联的基因组片段,然后用特异性抗体富集含有目的蛋白结合的基因组片段,并将目的DNA片段纯化后,进行建库测序。整个流程需要百万个细胞作为起始材料。此外,随着单细胞测序技术的发展,单细胞转录组已广泛应用,而单细胞水平表观机制的研究需求越来越多。 今天就和大家介绍下爱基百客的新产品——单细胞ChIP-seq技术。 01. 单细胞ChIP-seq技术的起源 2015年哈佛大学David Weitz院士团队首次提出了单细胞ChIP-seq技术——Drop-ChIP[1]。联合微流控、DNA barcoding和高通量测序来生成小鼠胚胎干细胞(ES)、胚胎成纤维细胞(mef)和造血祖细胞(EML细胞)H3K4me3和H3K4me2单细胞分辨率的染色质图谱。这个技术中用到的酶是微球菌核酸酶(MNase)。
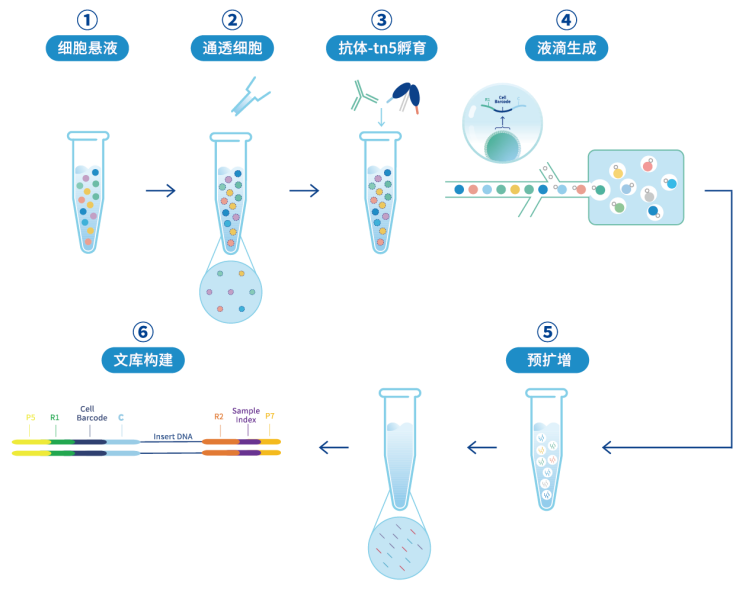
Drop-ChIP程序概述 随后,2019年有3个研究团队分别在高分期刊上发表单细胞ChIP-seq的方法文章。它们分别是CoBATCH、CUT&Tag和ACT-seq[2,3,4]。当前实验室中常用的单细胞ChIP-seq方法,主要就是这三种。核心技术都有用到刀豆蛋白偶联的磁珠、protein A-Tn5融合蛋白。技术上,这三种方法都有所改进,但仍有一些局限性,比如门槛高、人力成本高以及操作繁琐。 02. 爱基百客MobiChIP技术 爱基百客引入商业化的MobiNova®-100平台,一键式在6min内产生成千上万个单细胞的液滴。该技术平台在汲取前人优点的技术上,开发出一套全新的单细胞ChIP-seq解决方案。 技术路线:制备单细胞悬液,然后对细胞进行处理,增加细胞膜通透性,加入一抗去结合修饰位点,加入二抗扩大信号,加入PA-Tn5切割目的片段。随后,通过液滴微流控技术将经过处理的单细胞包裹形成油包水结构。进行预扩增,单个细胞的DNA被捕获和标记。最后,进行PCR扩增并加上接头序列,构建好文库即可上机测序。
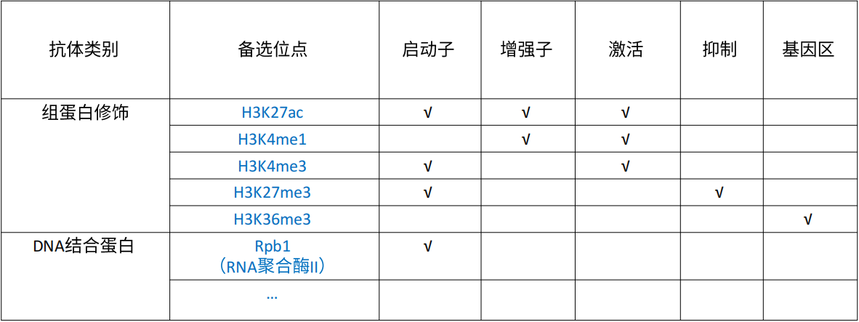
技术路线 已验证6大常用抗体(H3K27ac、H3K4me1、H3K4me3、H3K27me3、H3K36me3和Rpb1),满足绝大部分科研需求。
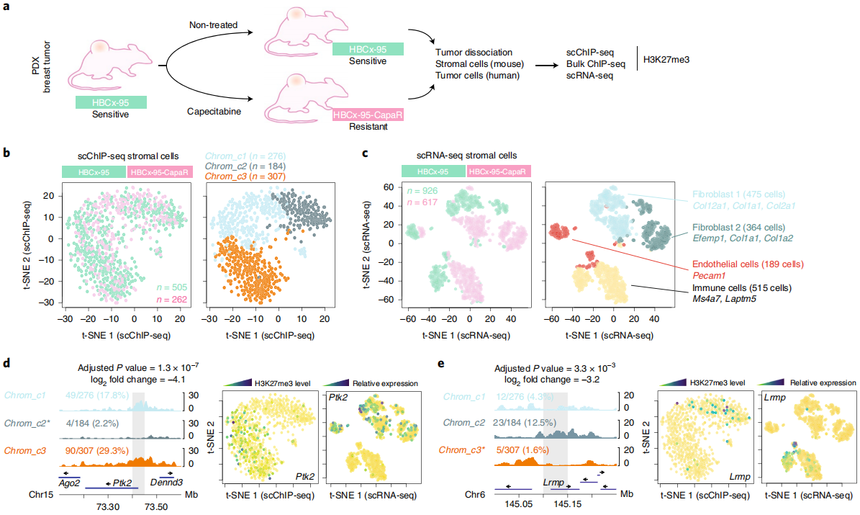
值得一提的是这6大常用抗体都曾经在组织层面发现广泛参与肿瘤疾病、器官发育、细胞分化等调控机制。比如: 1. H3K27ac主要位于活跃转录基因的启动子和增强子区域,在这些区域它与H3K4me3共存,共同促进基因激活表达。此外,H3K27ac还可在基因间区域形成超级增强子,进一步促进基因表达。 2. H3K4me1是活性增强子的标记,它和H3K27ac被广泛用于增强子基因组图谱的绘制。 3. H3K4me3也是热门组蛋白修饰之一,今年Nature发表的一篇研究报道H3K4me3可以调节RNA聚合酶II的移动控制基因表达[6]。 4. H3K27me3作为转录抑制标志被大家所熟知,在多种肿瘤中存在异常表达,在不同肿瘤中它的表达、功能及对预后的作用存在异质性。 03. 单细胞ChIP-seq的应用 尽管商业化的单细胞ChIP-seq技术近期才发布,但是单细胞ChIP-seq技术已在几个领域发光发热(比如肿瘤耐药性、细胞发育以及衰老)。 2019年一篇发表在期刊Nature genetics的研究利用单细胞ChIP-seq在乳腺癌获得性化疗耐药和靶向治疗的患者来源的异种移植模型[5],发现未经治疗的药物敏感肿瘤中的一部分细胞与耐药细胞具有共同的染色质特征,这是bulk ChIP-seq无法检测到的。这些细胞,以及来自耐药性肿瘤的细胞,失去了H3K27me3染色质标记,它与已知的促进治疗耐药性的基因的稳定转录抑制有关。该研究是基于单细胞ChIP-seq解析乳腺癌耐药性产生的基因调控机制。
图:小鼠基质细胞的单细胞ChIP-seq分析揭示了从H3K27me3染色质景观中获得的细胞特性 爱基百客在bulk ChIP-seq上积累丰富的项目经验,这些对于单细胞ChIP-seq是极大的助力,期待有相关需求的老师和我们沟通交流。此外,单细胞ChIP作为新产品,我们也送出了相应的福利活动。
[1] Rotem A, Ram O, Shoresh N, et al. Single-cell ChIP-seq reveals cell subpopulations defined by chromatin state. Nature Biotechnology. 2015. 33(11):1165-72. [2]Wang Q, Xiong H, Ai S, Yu X, Liu Y, Zhang J, He A. CoBATCH for High-Throughput Single-Cell Epigenomic Profiling. Mol Cell. 2019, 76(1):206-216. [3]Kaya-Okur HS, Wu SJ, Codomo CA, Pledger ES, Bryson TD, Henikoff JG, Ahmad K, Henikoff S. CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nat Commun. 2019, 10(1):1930. [4]Carter B, Ku WL, Kang JY, Hu G, Perrie J, Tang Q, Zhao K. Mapping histone modifications in low cell number and single cells using antibody-guided chromatin tagmentation (ACT-seq). Nat Commun. 2019, 10(1):3747. [5]Grosselin K, Durand A, Marsolier J, Poitou A, et al. High-throughput single-cell ChIP-seq identifies heterogeneity of chromatin states in breast cancer. Nature genetics. 2019, 51(6):1060-1066. [6]Wang H,Fan Z, Shliaha PV, et al. H3K4me3 regulates RNA polymerase II promoter-proximal pause-release. Nature. 2023. 616, E7.
市场部小助理,项目咨询 了 解 更 多 { 往 期 精 彩 回 顾 } 精选合集,欢迎收藏哟! |