NEWS
新闻资讯
|
DNA甲基化对植物生长发育到底有多重要?
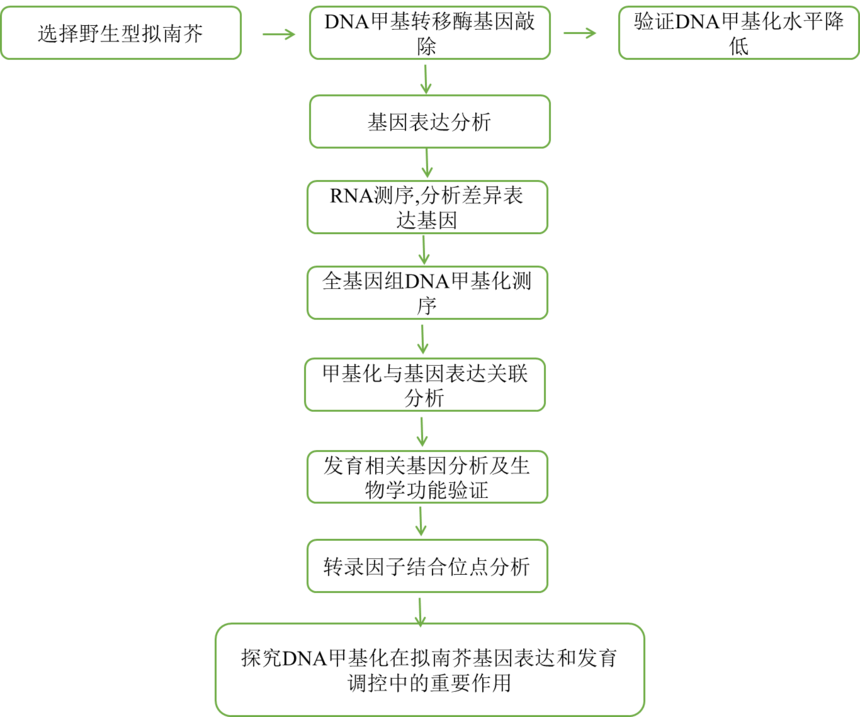
2022年3月14日,中国科学院分子植物科学卓越创新中心,上海植物逆境生物学研究中心的朱健康团队在Nature Communications杂志在线发表了题为“这篇文章旨在通过对DNA甲基化缺失的拟南芥植株进行全面的表达谱分析和发育研究,揭示DNA甲基化在植物中调控基因表达和发育的关键作用。通过深入了解DNA甲基化的功能,这项研究有望为植物生物学领域提供新的见解,也为未来利用基因编辑技术改良农作物品质和产量等方面的应用提供重要的理论基础。”的研究论文。该项研究首次创造无DNA甲基化的植物,全面揭示了DNA甲基化在植物发育和基因表达调控过程中的作用。 01 研究背景 胞嘧啶残基C-5位的DNA甲基化是一种保守的表观遗传修饰。在植物中,DNA甲基化发生在CG、CHG和CHH(H表示A、T或C)三种碱基序列的胞嘧啶上[1,2,3]。CG甲基化由DNA甲基转移酶MET1维持,CHG甲基化由CMT3和CMT2维持,CHH甲基化则由CMT2和DRM1/2维持[4]。尽管这五种DNA甲基转移酶的作用机制已被广泛研究,但它们是否足以维持整个基因组的 DNA甲基化尚不清楚。为了解决这一问题,最近的研究采用了一种全新的方法,成功地构建了一个DNA甲基化缺失的拟南芥植株。通过基因编辑技术,研究人员能够精确地靶向DNA甲基化酶基因,从而使植株在没有DNA甲基化的情况下生长和发育。这样的研究材料为科学家提供了一个理想的模型,可以直接研究DNA甲基化在植物基因表达和发育中的功能。 02 研究思路
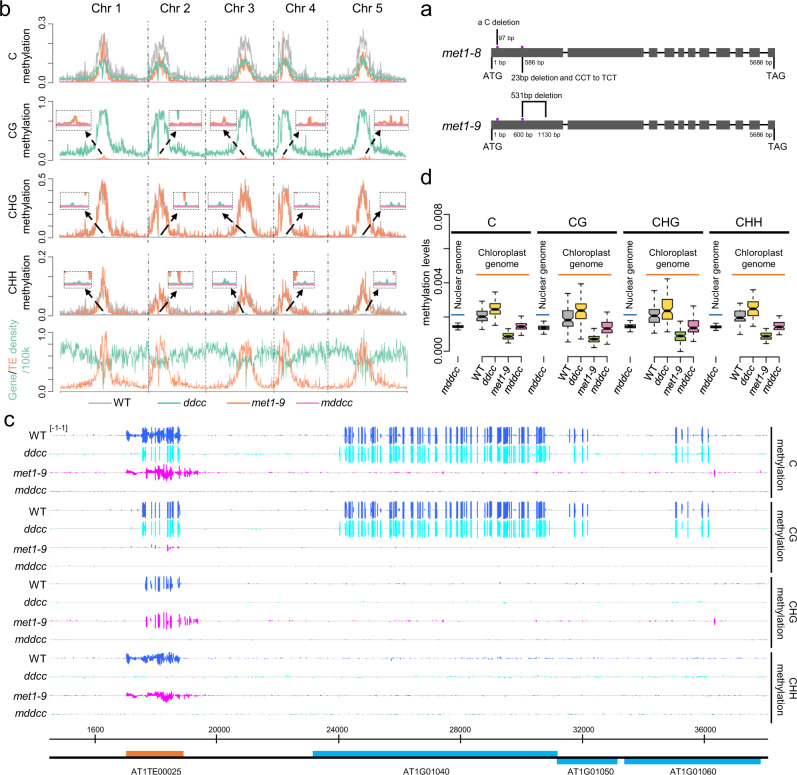
03 研究结果 一、MET1,DRM1,DRM2,CMT3和CMT2维持拟南芥基因组的DNA甲基化 为了确定五种已知的DNA甲基转移酶是否维持拟南芥植物的整个DNA甲基化,作者将CRISPR / Cas9介导的基因编辑与传统遗传学相结合,敲除相应的蛋白质编码基因[5]。首先,将cmt2与drm1 - drm2 - cmt3 (ddc)三突变体杂交,分离出drm1 - drm2 - cmt3 - cmt2 (ddcc)四突变体;随后使用带有两个sgRNAs的CRISPR/Cas9系统靶向MET1的第一个外显子,在ddcc背景下产生MET1突变。在T2代获得了不含Cas9的met1/+ drm1 drm2 cmt3 cmt2种子,并从这些植物的后代中分离到met1 drm1 drm2 cmt3 cmt2 (mddcc)个体。将四重突变背景中产生的met1等位基因命名为met1-8(图1a)。与此同时,同样的CRISPR/Cas9载体被用于产生一个met1单突变体;在WT背景下产生的等位基因命名为met1-9(图1a)。 为了检测ddcc,met1-9和mddcc突变体中的DNA甲基化水平,作者进行了全基因组亚硫酸氢盐测序,结果发现met1-9突变体中几乎没有CG甲基化,ddcc突变体中几乎没有非CG甲基化(图1b-c)。对各染色体DNA甲基化水平的分析发现,相对于mddcc,在met1-9和ddcc的周围区域分别保持低水平的CG和非CG甲基化,表明MET1和DRM1 / DRM2 / CMT2 / CMT均可以在一定程度上维持非CG和CG甲基化(图1b)。为了研究mddcc突变体中的DNA甲基化是否完全消除,作者将核基因组的DNA甲基化水平与无甲基化的叶绿体基因组的DNA甲基化水平进行了比较。值得注意的是,在三种序列中,mddcc核基因组DNA甲基化与叶绿体基因组一样低(图1d),表明MET1,DRM1,DRM2,CMT3和CMT2负责维持拟南芥基因组中DNA甲基化。因此,mddcc突变体是无DNA甲基化的拟南芥基因型。
图1 DNA甲基化在mddcc突变体中被消除 二、DNA甲基化以剂量效应的方式调节基因表达 作者对WT、ddcc、met1-9和mddcc进行了RNA-seq,结果发现,与WT相比,met1-9中有几千个差异基因,而ddcc中只有几百个差异基因,这表明CG甲基化对基因转录的直接和间接影响远强于非CG甲基化。此外,mddcc突变体的差异基因数量明显高于met1-9,表明CG和非CG甲基化在基因表达调控中存在部分功能冗余(图2a)。ddcc、met1-9和mddcc中具有多个交集基因(图2b-c)。 为了更详细地分析DNA甲基化在基因表达调控中的功能,作者首先根据WT背景下甲基化水平将17930个可检测到表达的基因分为五大类:3463个(19.3%)基因仅在CG中发生基因体甲基化(gbM基因),292个(1.6%)基因在非CG环境中发生基因体甲基化(teM基因),3773个(21.0%)基因在其启动子中发生甲基化(pM基因),2367个(13.2%)基因在其3 '下游区域发生甲基化(dM基因),8035个(44.8%)基因未甲基化(UM基因)(图2d)。差异基因在gbM基因中所占比例较低。在dM基因中存在一定比例的差异基因,这表明基因3 '下游的DNA甲基化在调节基因表达中也发挥了作用。teM基因中上调基因比例显著高于下调基因比例,说明非CG基因体甲基化在基因抑制中起主要作用。此外,在gbM、pM和dM亚群中可以鉴定出相似数量的上调和下调基因(图2f)。综上所述,研究结果证实了DNA甲基化对基因表达的抑制作用,但也暗示了DNA甲基化的广泛激活功能。 有趣的是,通过热图比较ddcc、met1-9和mddcc突变体中相同DEGs的基因表达水平(图2b、c、e),结果发现甲基化基因(gbM、teM、pM和dM)中的DNA甲基化以多种方式影响基因表达。根据不同突变背景下DEG行为,可以区分DNA甲基化对基因表达的影响如下:(1)冗余性,即仅在mddcc中表达增加/减少;(2)剂量I,在CG甲基化缺失时即met1-9中表达轻度增加/减少,在非CG甲基化缺失(mddcc)后表达进一步增加/减少;(3)剂量II,在非CG甲基化缺失时即ddcc中表达轻度增加/减少,在CG甲基化缺失(mddcc)后表达进一步增加/减少;(4)剂量III,当CG或非CG甲基化缺失时(met1-9或ddcc)表达微弱增加/减少,在DNA甲基化完全缺失时(mddcc)表达进一步增加/减少;(5)mCG沉默/激活,当met1-9中的基因的上调/下调程度与mddcc相当时,被mCG沉默/激活;(6)非mCG沉默,当ddcc和mddcc中的基因上调程度相似时,被非mCG沉默(图2e)。 受剂量效应调控的基因数量明显高于其它调控方式的基因数量(图2g),这表明DNA甲基化主要以剂量依赖的方式调节基因表达。与最常见的剂量效应剂量I调控相比,剂量II和剂量III的情况较少(图2g),这表明在正常情况下,CG甲基化在调节基因表达中起主要作用,而非CG甲基化的主要功能是在CG甲基化缺失时防止基因错误表达。值得注意的是,与之前的结果一致,met1-9中甲基化DEG的CHG和CHH甲基化相对于WT轻度降低。因此,非CG甲基化的变化也可能导致met1-9中检测到的基因表达的变化。 值得注意的是,ddcc、met1-9和mddcc中很大比例DEG是UM基因(图2h)。这些基因的表达变化可能是由甲基化基因的表达变化间接引起的,尽管不能排除这些基因的差异表达是由于微量DNA甲基化水平的丧失造成的(图2e)。
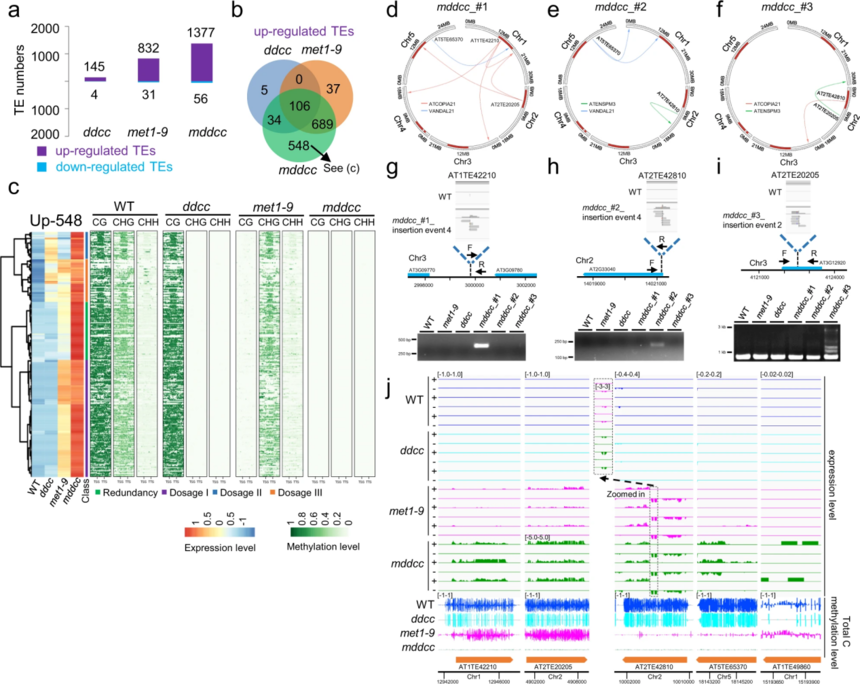
图2 DNA甲基化以剂量效应的方式调节基因表达 三、CG和非CG甲基化共同抑制TE的表达和转座 作者在WT与突变体中鉴定出DETs,正如预期一样,大多数TE在这些突变体中上调,这也证实了CG甲基化在抑制TE表达中起主要作用,非CG甲基化与CG甲基化冗余抑制一部分TE(图3a)。与前面观察结果一致,DNA甲基化对TE表达的影响表现出多种模式,包括冗余、剂量I、剂量II、剂量III、mCG沉默和非mCG沉默(图3b-c)。因此,CG和非CG甲基化以各种方式共同抑制TE表达,DNA甲基化与TE表达之间存在剂量效应关系。 为了研究这些突变体中TE再激活是否可能导致TE转座,作者对WT、ddcc、met1-9和mddcc突变体进行了DNA测序,结果显示在ddcc、met1-9和mddcc中均发现了假定的TE转座事件;然而,只有11个TE转座事件可以通过PCR证实,全部发生在mddcc中(图3d-i)。11个转位事件涉及5个TEs,其中AT1TE42210、AT2TE20205、AT5TE65370和AT1TE49860在mddcc中表达水平最高,表明TE的高转录激活程度仅与转座激活部分相关。与先前的结果一致,MET1突变仅在连续自交后才导致有限数量的TE转座,在CRISPR-Cas9新产生的met1 -9突变体中未发现任何TE。值得注意的是,在mddcc突变体的第一代纯合子中发现了11个转座事件。该研究结果表明CG和非CG甲基化冗余地阻止TEs转座。
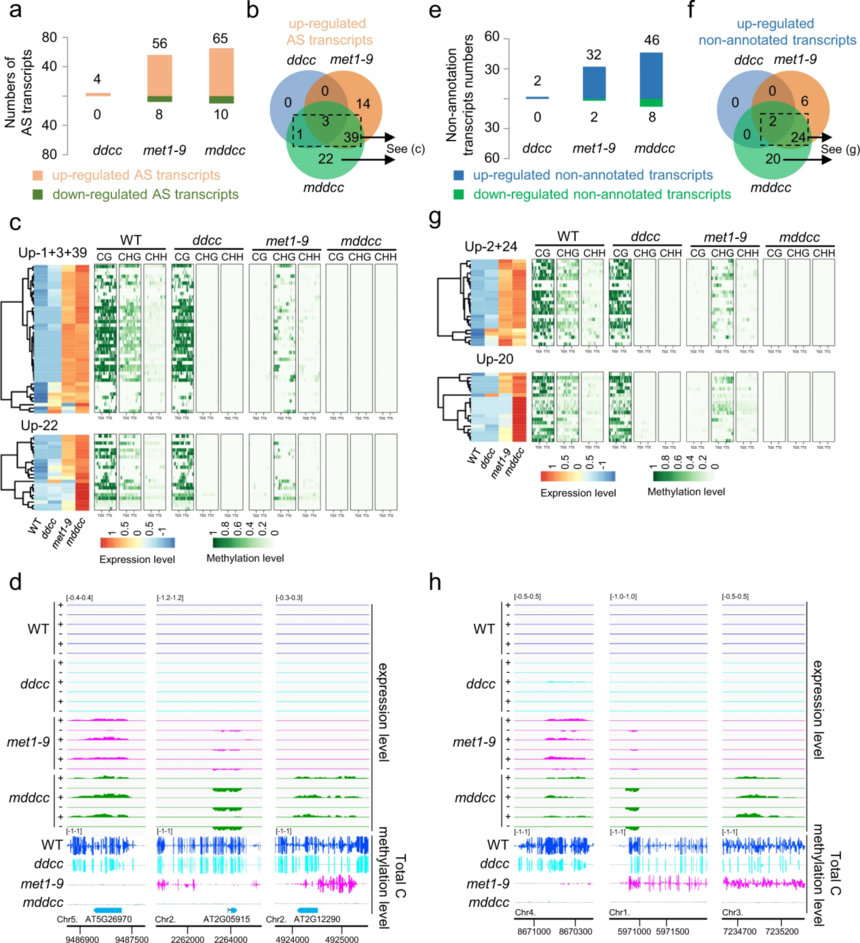
图3 CG和非CG甲基化共同抑制TE的激活和转座 四、DNA甲基转移酶缺失的突变体中反义转录物和无注释转录物的表达 基因体中的DNA甲基化被认为可以抑制基因内反义转录物的表达;然而,MET1突变会导致基因体甲基化的大量缺失,只会导致少数基因内反义转录物的过度表达。作者假设CG和非CG甲基化可能冗余地抑制基因内反义转录物的表达。为了验证这一观点,分析发现与WT相比,ddcc、met1-9和mddcc突变体中的反义转录物表达显著改变。在met1-9和mddcc突变体中,错误表达的反义转录物数量较低,但高于ddcc突变体。与met1-9相比,mdcc中差异表达的反义转录本数量增加较少(图4a-b)。正如预期的那样,在WT植物中,大多数上调表达的反义转录物的基因均发生大量DNA甲基化(图4c-d)。这些结果支持基因体中CG和非CG甲基化在有限数量位点上的抑制反义转录方面起作用。 此外,作者在mddcc突变体中发现了46个无注释转录元件(图4e-f)。在WT植物中,绝大多数上调的无注释转录本位点显示出密集的DNA甲基化(图4g-h)。这一发现表明,拟南芥基因组中存在其他转录元件,其表达被DNA甲基化阻断。
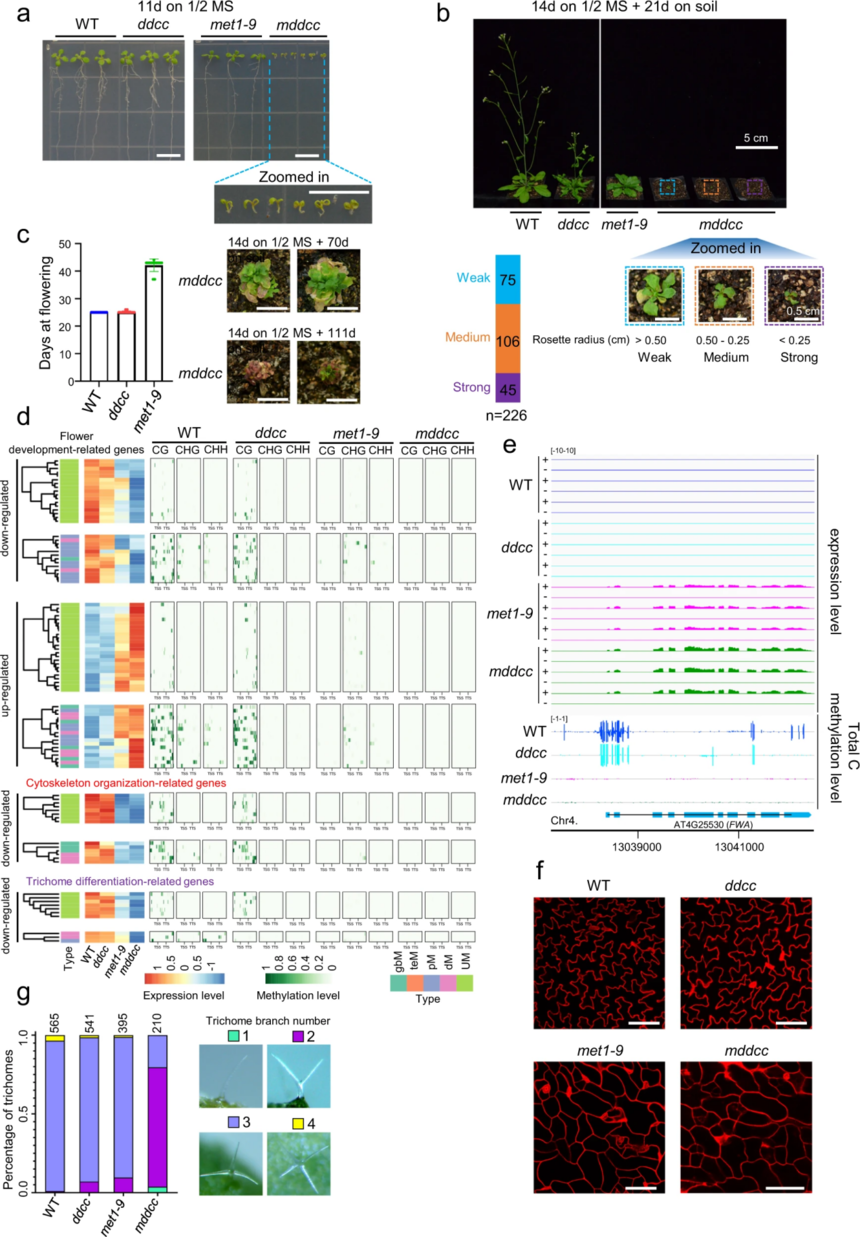
图4 DNA甲基化抑制反义转录本和无注释转录本的表达 五、DNA甲基化是拟南芥正常发育所必需的,但对其生存不是必要的 为了研究DNA甲基化对植物发育的意义,作者对ddcc、met1-9和mddcc突变体的整个生命周期进行了表型分析。ddcc和met1-9的幼苗大小与WT相似,而mddcc的幼苗非常小(图5a)。当mddcc在土壤中生长时,矮小表型更加明显(图5b)。这种表型变异可能是由这些植物中的TE转座引起的(图3)。值得注意的是,尽管mddcc植株生长缓慢,但在长日照条件下,它们的存活时间比其他基因型要长,由此得出结论,DNA甲基化对拟南芥的发育是必要的,但对其存活是非必要的。然而,作者发现mddcc突变体从未开花(图5c),这可能与花发育相关基因的异常表达有关(图5d)。先前研究表明,在met1突变体中,FWA启动子DNA甲基化的缺失会导致FWA异位表达,从而导致晚开花。由于FWA的表达水平在met1-9和mddcc之间具有可比性(图5e),因此这两个突变体之间的表型差异一定是由其他基因造成的。 有趣的是,MET1的突变足以将铺板细胞的形状改变为规则形状,并导致自发性细胞死亡。与此一致的是,在控制铺板细胞形状中起关键作用的细胞骨架组织相关基因和细胞死亡相关基因在met1-9和mddcc突变体中分别显著下调和上调表达(图5d)。WT、ddcc和met1中毛状体通常有三个分支,而mddcc中大多数毛状体有两个分支(图5),表明CG和非CG甲基化冗余地促进了毛状体的发育。DNA甲基化对植物的维管发育至关重要,研究发现mddcc突变体的维管系统显著性减少。基因表达分析验证了这一猜想,因为参与毛状体分化和维管发育的多个基因在mddcc中显著的异常表达。鉴于mddcc突变体的极端发育缺陷,DNA甲基化是否可能是分生组织活性所必需的。met1-9和ddcc突变体的茎尖分生组织与WT相当,但mddcc突变体的SAM严重减少,这意味着CG和非CG甲基化冗余参与了SAM的发育。
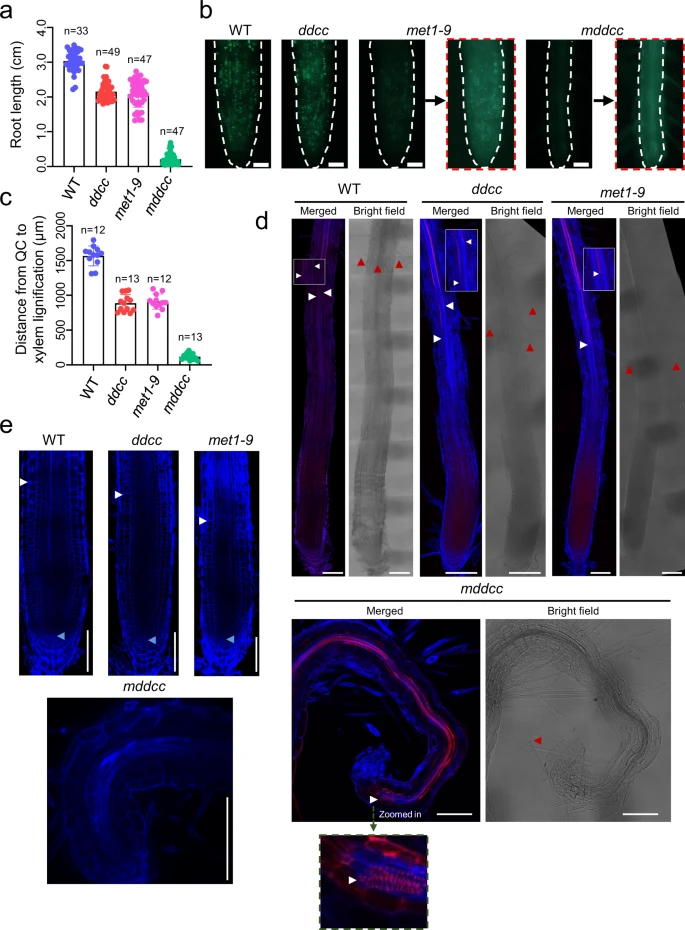
图5 mddcc突变体表现出一系列极端的发育缺陷 与WT相比,ddcc和met1的根长度略有减少,而mddcc的根长度则急剧减少(图6a)。为了检测RAM的活性,作者使用胸腺嘧啶类似物5-乙基-2 ' -脱氧尿嘧啶(EdU)来观察该区域的细胞增殖。分析发现,作为DNA复制和细胞分裂的代表,EdU信号在met1-9中显著减少,而在mddcc中几乎检测不到(图6b),这表明CG甲基化有助于RAM中的细胞增殖,非CG甲基化部分冗余。值得注意的是,在mddcc中木质部分化和根毛分化发生得早,经常直接在根尖观察到(图6c-e);这种表型表明CG和非CG甲基化冗余地抑制根细胞分化和/或促进分生组织维持。
图6 CG和非CG甲基化共同调节RAM的活性 因此,这篇文章旨在通过对DNA甲基化缺失的拟南芥植株进行全面的表达谱分析和发育研究,揭示DNA甲基化在植物中调控基因表达和发育的关键作用。通过深入了解DNA甲基化的功能,这项研究有望为植物生物学领域提供新的见解,也为未来利用基因编辑技术改良农作物品质和产量等方面的应用提供重要的理论基础。同时,这项研究还有望对人类的健康产生一定的启示,因为DNA甲基化在动植物细胞中都起着重要的调控作用,对许多疾病的发病机制也可能产生影响。综上所述,这篇文章的研究成果将有助于推动我们对DNA甲基化调控机制的全面认识,为植物生物学和相关领域的研究提供新的突破。 爱基百客专注于提供领先的表观组学服务,可提供方案设计、样本制备、测序、分析以及验证一站式服务。有相关需求的老师欢迎联系区域销售。
市场部小助理,项目咨询 参考文献 [1]. Lindroth, A. M. et al. Requirement of CHROMOMETHYLASE3 for maintenance of CpXpG methylation. Science 292, 2077–2080 (2001). [2]. Du, J. et al. Dual binding of chromomethylase domains to H3K9me2-containing nucleosomes directs DNA methylation in plants. Cell 151, 167–180 (2012). [3]. Stroud, H. et al. Non-CG methylation patterns shape the epigenetic landscape in Arabidopsis. Nat. Struct. Mol. Biol. 21, 64–72 (2014). [4].Zemach, A. et al. The Arabidopsis nucleosome remodeler DDM1 allows DNA methyltransferases to access H1-containing heterochromatin. Cell 153,193–205 (2013). [5]. Zhang, Z. et al. A multiplex CRISPR/Cas9 platform for fast and efficient editing of multiple genes in Arabidopsis. Plant Cell Rep. 35, 1519–1533 (2016). 了 解 更 多 { 往 期 精 彩 回 顾 } 精选合集,欢迎收藏哟! |