NEWS
新闻资讯
|
国自然热点|超级增强子“super”在哪?cell重磅发现:新型DNA调控元件——促进子
增强子(enhancer),又可称为强化子,是DNA上一段可与蛋白质(反式作用因子,trans-acting factor)结合的区域,可以被转录因子等蛋白结合从而激活基因转录。1981年,增强子首次被描述为猿猴病毒40(SV40)基因组中一个72bp的重复序列,可以使报告基因的异位表达增加约200倍。1983年,在哺乳动物的小鼠免疫球蛋白重链基因中发现了增强子。随后,在各种细胞和组织中有不同的增强子被报道[1]。 超级增强子(super enhancer,SE)是一类由多个增强子(增强子间间隔小于12.5kb)组成的一簇增强子,通常认为超级增强子比普通增强子的调控能力更强,在机体调控过程中起到更重要的作用。作为目前的国自然研究热点,超级增强子仍然存在许多疑点:超级增强子仅仅是一些传统增强子的集合,还是这些增强子相互之间具有协同作用【2-4】?是否集合中每个元件都起到了增强子的作用?为何删除一个或者多个增强子的研究方法会得到相互矛盾的结论【5-11】? 2023年12月14日,英国约翰·拉德克利夫医院的Mira Kassouf和Douglas R. Higgs团队联合在Cell杂志上发表了文章Super-enhancers include classical enhancers and facilitators to fully activate gene expression。作者提出了一个新概念:促进子。促进子没有内在的增强子活性,但在其缺失的情况下,经典增强子上调靶基因的功能会被减弱。本文为进一步研究超级增强子的调控机制提供了一个新的方向,值得各位研究。
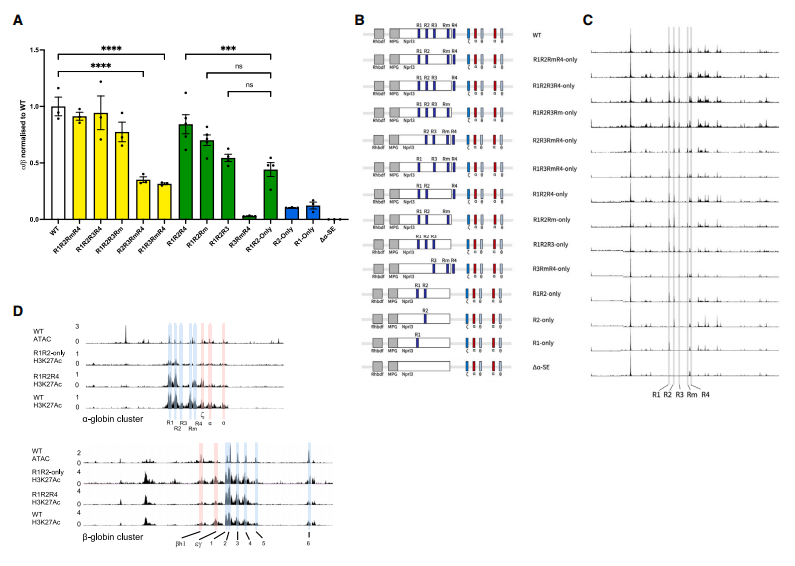
小鼠α-globin SE (α-globin SE,α-SE)由5个组成元素(R1、R2、R3、Rm和R4)组成;它与重复的α-globin基因一起位于一个明确的65kb亚拓扑相关域(sub-TAD)中,并在终末分化的红细胞中上调α-globin基因的表达。其中,R1和R2是能够显著上调基因表达的经典增强子,而R3、Rm和R4则是非活性元件。研究团队通过基因编辑的手段,设计了(1)含有野生型(WT)α-globin亚TAD;(2)只保留R2的等位基因(R2-only);(3)将α-SE所有元件都删除,创建无增强模型(Da-SE);(4)仅删除R2(△R2)。 作者发现,删去R2会降低α-globin转录活性的50%,但仅含有R2的细胞种,α-globin的转录水平只有WT的10%左右,与前期推测的R2负责α-SE所有转录活性的50%的结论截然不符,这暗示着其它几个元件的存在对于α-globin的表达有着超出预期的作用。 随后,作者通过ATAC-seq、ChIP-seq、3C、RNA-seq等测序实验,发现,R2-only中,R2和α-globin的启动子区可及性、H3K4Me1、H3K4Me3以及H3K27Ac修饰依然存在,但与WT相比明显降低。此外,增强子招募Mediator复合物的能力大幅下降,这同样导致Pol II在α-globin启动子区的结合大幅下降。
图:生成一个无增强子(△α-SE)体外小鼠模型和一个仅含R2的体内小鼠模型,以测试R2增强子元件的充分性 为了研究其他元件在调控中可能存在的机制,作者构建了仅存在R1和R2的突变体,并分别导入R3、Rm和R4。发现,三个辅助元件的导入不同程度上提高了α-globin的表达,并且重新导入R4促进了H3K27ac的修饰水平。此外,R3和R4的位置也会影响基因表达(距离α-globin启动子更近能有效提高基因表达)。
图:R1和R2增强子依赖于R3、Rm和R4来充分发挥其潜力 基于此,作者利用合成生物学、基因编辑,并结合多种表观测序手段(ATAC-seq/ChIP-seq/3C等),发现了超级增强子中一种新的调控机制,并命名为促进子。此外,作者认为,该研究中描述的促进子可能是多元件超级增强子中相对常见的组成元件,对促进子进行鉴定以及工作原理研究很可能会对基因的正常开启/关闭机制,以及在人类遗传疾病中这种机制是如何出错的提供新的见解。 文章链接:DOI:https://doi.org/10.1016/j.cell.2023.11.030 PS:此篇文章中作者使用的ATAC-seq、ChIP-seq、RNA-seq等多种测序技术爱基百客也能提供,欢迎各位老师咨询。此外,我们也针对超级增强子方向提供了一个可行性方案,感兴趣的老师可以联系销售获取~
项目咨询
一文了解国自然热点“超级增强子”的重要标记——H3K27ac 【调控组文献分享】玉米中的DNA和染色质特征测序绘制全基因组增强子图谱
[1] Blackwood EM, Kadonaga JT. Going the distance: acurrent view of enhancer action. Science, 1998,281(5373):60–63. [2] Blobel, G.A., Higgs, D.R., Mitchell, J.A., Notani, D., and Young, R.A. (2021). Testing the super-enhancer concept. Nat. Rev. Genet. 22, 749–755. [3] Grosveld, F., van Staalduinen, J., and Stadhouders, R. (2021). Transcriptional Regulation by (Super)Enhancers: From Discovery to Mechanisms. Annu. Rev. Genom. Hum. Genet. 22, 127–146. [4] Moorthy, S.D., Davidson, S., Shchuka, V.M., Singh, G., Malek-Gilani, N., Langroudi, L., Martchenko, A., So, V., Macpherson, N.N., and Mitchell, J.A. (2017). Enhancers and super-enhancers have an equivalent regulatory role in embryonic stem cells through regulation of single or multiple genes. Genome Res. 27, 246–258. [5] Bender, M.A., Ragoczy, T., Lee, J., Byron, R., Telling, A., Dean, A., and Groudine, M. (2012). The hypersensitive sites of the murine b-globin locus control region act independently to affect nuclear localization and transcriptional elongation. Blood 119, 3820–3827. [6] Hay, D., Hughes, J.R., Babbs, C., Davies, J.O.J., Graham, B.J., Hanssen, L., Kassouf, M.T., Marieke Oudelaar, A.M., Sharpe, J.A., Suciu, M.C., et al.(2016). Genetic dissection of the a-globin super-enhancer in vivo. Nat.Genet. 48, 895–903. [7] Hnisz, D., Schuijers, J., Lin, C.Y., Weintraub, A.S., Abraham, B.J., Lee, T.I.,Bradner, J.E., and Young, R.A. (2015). Convergence of Developmental and Oncogenic Signaling Pathways at Transcriptional Super-Enhancers.Mol. Cell 58, 362–370. [8] Ho¨ rnblad, A., Bastide, S., Langenfeld, K., Langa, F., and Spitz, F. (2021).Dissection of the Fgf8 regulatory landscape by in vivo CRISPR-editing reveals extensive intra- and inter-enhancer redundancy. Nat. Commun.12, 439. [9] Huang, J., Li, K., Cai, W., Liu, X., Zhang, Y., Orkin, S.H., Xu, J., and Yuan,G.-C. (2018). Dissecting super-enhancer hierarchy based on chromatin interactions. Nat. Commun. 9, 943. [10] Shin, H.Y., Willi, M., HyunYoo, K., Zeng, X., Wang, C., Metser, G., and Hennighausen, L. (2016). Hierarchy within the mammary STAT5-driven Wap super-enhancer. Nat. Genet. 48, 904–911. [11] Thomas, H.F., Kotova, E., Jayaram, S., Pilz, A., Romeike, M., Lackner, A.,Penz, T., Bock, C., Leeb, M., Halbritter, F., et al. (2021). Temporal dissection of an enhancer cluster reveals distinct temporal and functional contributions of individual elements. Mol. Cell 81, 969–982.e13 了 解 更 多 { 往 期 精 彩 回 顾 } 精选合集,欢迎收藏哟! |