NEWS
新闻资讯
|
Nucleic Acids Research | scATAC-seq+CUT&Tag探究关键转录因子对视网膜细胞分化的调控作用
在中枢神经系统发育过程,多能神经祖细胞如何产生不同的神经细胞类型仍然知之甚少。最近的scRNA-seq研究已经描绘了包括神经视网膜在内的许多神经系统中单个神经细胞类型的发育轨迹。进一步了解神经细胞多样性的形成需要了解表观遗传景观如何沿着个体细胞谱系变化以及关键转录因子如何调节这些变化。 2023年1月Nucleic Acids Research(IF:14.9)在线发表了题为“Key transcription factors influence the epigenetic landscape to regulate retinal cell differentiation”的研究论文,该研究利用scATAC-seq和CUT&Tag技术研究了视网膜细胞分化过程中的表观遗传景观变化,并找到了调控视网膜细胞分化的关键转录因子。
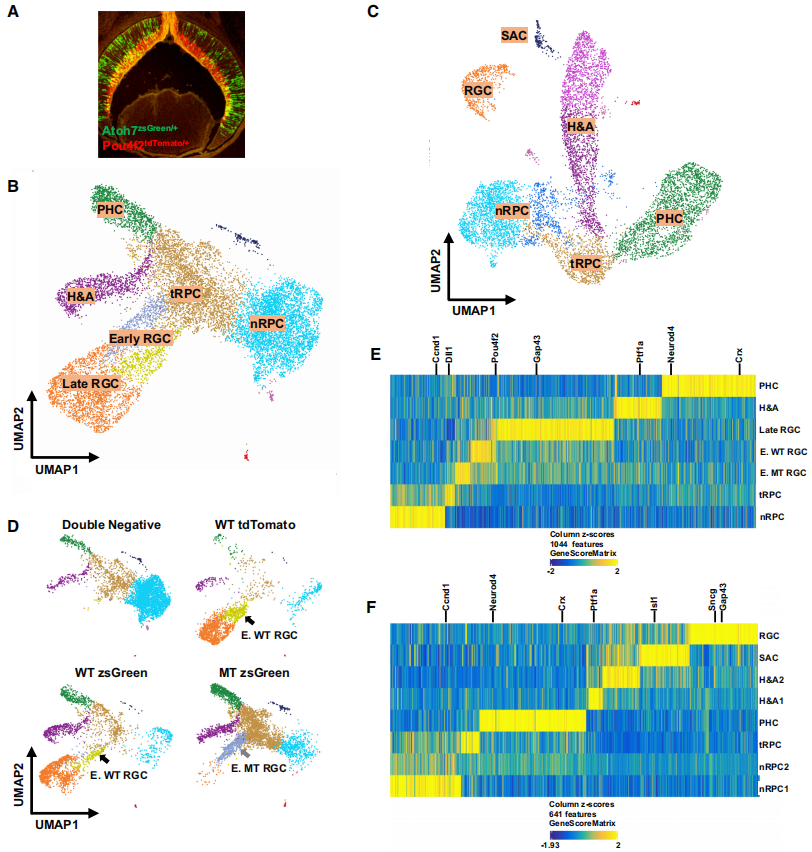
1. scATAC-seq识别与scRNA-seq相同的细胞状态/类型 为了探究视网膜细胞发育轨迹中表观遗传景观的变化,通过荧光激活细胞分选(FACS),利用Atoh7zsGreen(零等位基因)和Pou4f2tdTomato(野生型等位基因),分选出不同细胞状态的细胞(Fig1A)。收集了包括不同基因型的视网膜细胞样本,生成了scATAC-seq文库,并进行了测序。通过分析插入位点和识别重复峰,得到了E14.5和E17.5细胞样本的插入数据。这些数据揭示了在这两个发育阶段活跃的启动子和调控元件。使用ArchR进行维度缩减聚类分析,并通过UMAP显示了E14.5和E17.5细胞的聚类结果。结果显示E14.5细胞有7个聚类,E17.5细胞有8个聚类(Fig1B,1C)。通过GeneScore模型进行基因活性分析,确定了每个聚类在两个阶段的特异性基因模式。在E14.5聚类中包括nRPCs、tRPCs、H&As、PHCs、早期RGCs(WT和MT)和晚期RGCs(Fig1B,1D,1E);在E17.5聚类中包括nRPCs、tRPCs、H&As、PHCs和RGCs(Fig1C,1F)。通过FACS纯化的细胞样本对不同聚类的贡献支持了对聚类的身份分配(Fig1D)。UMAP预测的聚类结果和拓扑关系与先前的scRNA-seq聚类结果一致,表明scATAC-seq数据重现了早期视网膜发育中RGCs、H&As和PHCs的发育轨迹。E17.5的RGC聚类与其他聚类分离,并且没有早期RGC聚类,这与先前的scRNA-seq结果一致,表明在E17.5时RGC的发生已基本完成。总的来说,收集的细胞样本充分代表了这两个发育阶段的所有细胞群。
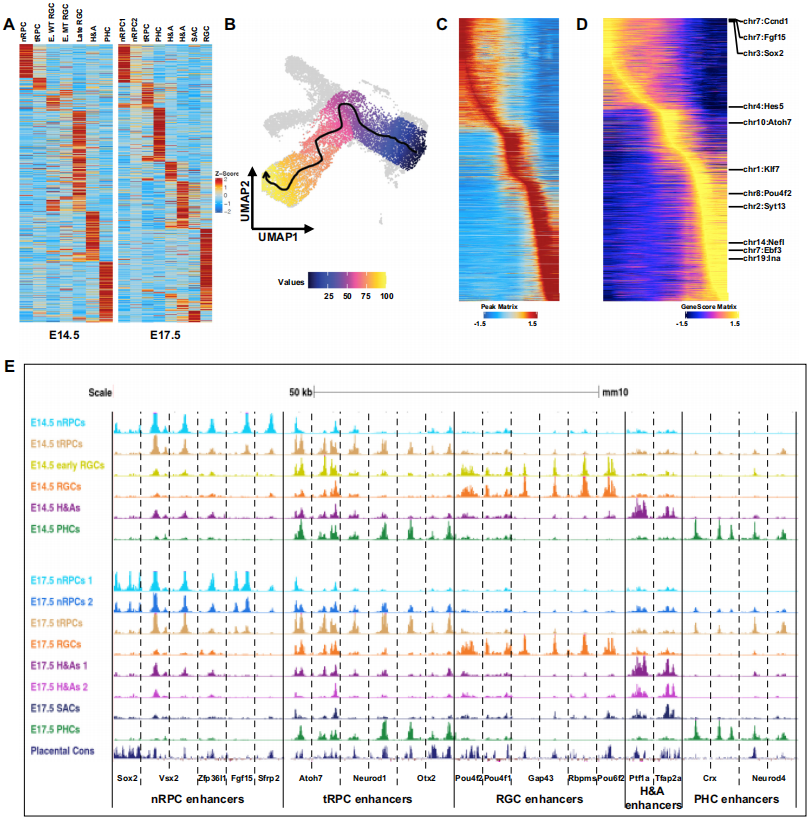
Fig 1.E14.5和E17.5视网膜细胞的scATAC-seq结果 2. 差异peak峰揭示了随个体发育轨迹变化的表观遗传景观 通过scATAC-seq对小鼠视网膜细胞进行了分析,发现不同的细胞群集由不同的差异可及峰定义,代表潜在的细胞状态/类型特异性增强子。在E14.5和E17.5样本中分别识别出了127,302个和151,719个差异可及峰,并为每个E14.5和E17.5群集确定了独特的差异可及峰集。基于这些峰的Z分数聚类进一步验证了这些群集之间的关系,揭示了从nRPCs到tRPCs,再到包括H&As、PHCs和RGCs在内的不同细胞命运的发育轨迹(Fig2A)。通过轨迹分析进一步确认了视网膜RGC谱系的表观遗传景观的转变,从nRPCs到tRPCs,再到早期RGCs,最终到更成熟的(晚期)RGCs(Fig2B,2C)。基因活动、细胞状态/类型特异性增强子以及基因表达的变化紧密相关,揭示了随着细胞分化的进行,表观遗传景观的转变(Fig2D、2E)。从E14.5到E17.5的时间变化也被研究,结果显示了所有细胞状态/类型的表观遗传景观的转变。研究结果为进一步理解RPCs的能力变化、产生的细胞类型的转变和早期生成的神经元的成熟提供了有用的信息。
Fig2. 变化的表观遗传景观是由视网膜细胞状态/类型特异性增强子定义的 3.差异活性增强子调节细胞状态/类型特异性基因 通过对E14.5和E17.5细胞群体进行的scRNA-seq和scATAC-seq数据整合,识别了与6600个基因相关的55293个P2G链接和8531个基因相关的76079个链接。多个增强子参与调控同一基因,且增强子的可及性与其调控基因的表达水平高度相关。从E14.5和E17.5两组数据的热图中可以看出,峰的可接近性或相应增强子的活性,以及它们所连接的基因的表达水平是高度相关的(Fig.3A,3B)。与先前通过scRNA-seq鉴定的细胞状态富集基因集进行比较后发现,84.5%和90.3%的基因分别包含在E14.5和E17.5基因集中(Fig.3C)。P2G分析有助于识别潜在增强子、调控基因及评估它们在这两个发育阶段所有细胞状态/类型中的活动。通过比较Atoh7和Otx2基因的候选增强子来评估其有效性。发现Atoh7有一个近端增强子和一个远端影子增强子,它们在不同细胞状态/类型中的活性动态反映了Atoh7表达的变化。而Otx2的增强子主要在tRPCs和/或PHCs中活跃,这些增强子与Otx2表达的动态密切相关(Fig.3D,3E)。研究结果表明,细胞状态/类型特异性增强子是细胞状态/类型特异性表达的主要驱动因素。
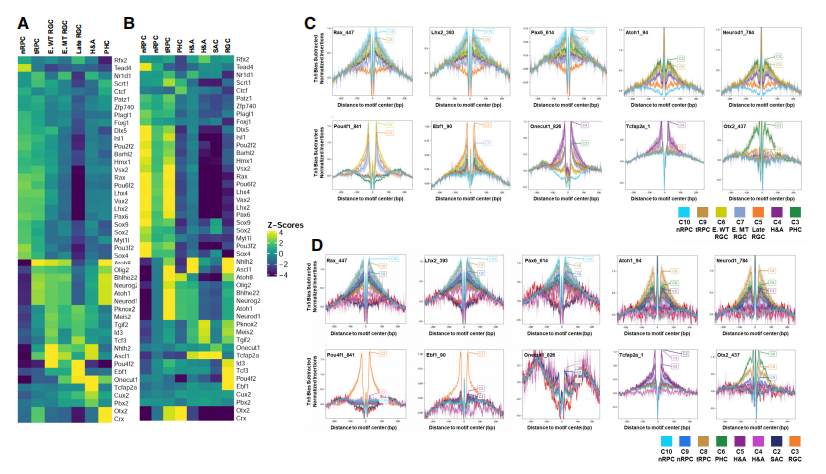
Fig3. P2G连锁分析在全基因组范围内确定单个基因的增强子 4. 转录因子在不同细胞状态/类型中起作用 许多在视网膜细胞分化不同阶段表达和功能的转录因子与研究者鉴定的细胞状态/类型特异增强子相互作用。不同E14.5和E17.5簇中富集了独特的DNA结合位点,许多由已知在相应细胞状态/类型中发挥功能的转录因子识别的DNA结合位点(Fig4A,4B)。邻近细胞状态倾向于共享富集的DNA结合位点,进一步突显了沿不同谱系发育轨迹的细胞状态之间的延续和转变。为了进一步验证以上所指的转录因子确实以细胞状态/类型特定的方式结合增强子,研究者还根据scATAC-seq数据进行了足迹分析,并观察这些序列图案的细胞状态/类型(Fig4C,4D)。在E14.5和E17.5,不同转录因子在不同细胞群中留下特异性足迹,表明这些转录因子在不同发育阶段和细胞类型中发挥作用。例如,Pou4f在早期和晚期RGCs中留下足迹,而Onecut因子在E14.5时在所有三个谱系中足迹显著,但在E17.5时在这些谱系中明显减少。这些发现表明,关键转录因子确实以细胞状态/类型特异方式结合增强子,参与塑造特定状态的表观遗传景观,并推动其转变到下一状态。
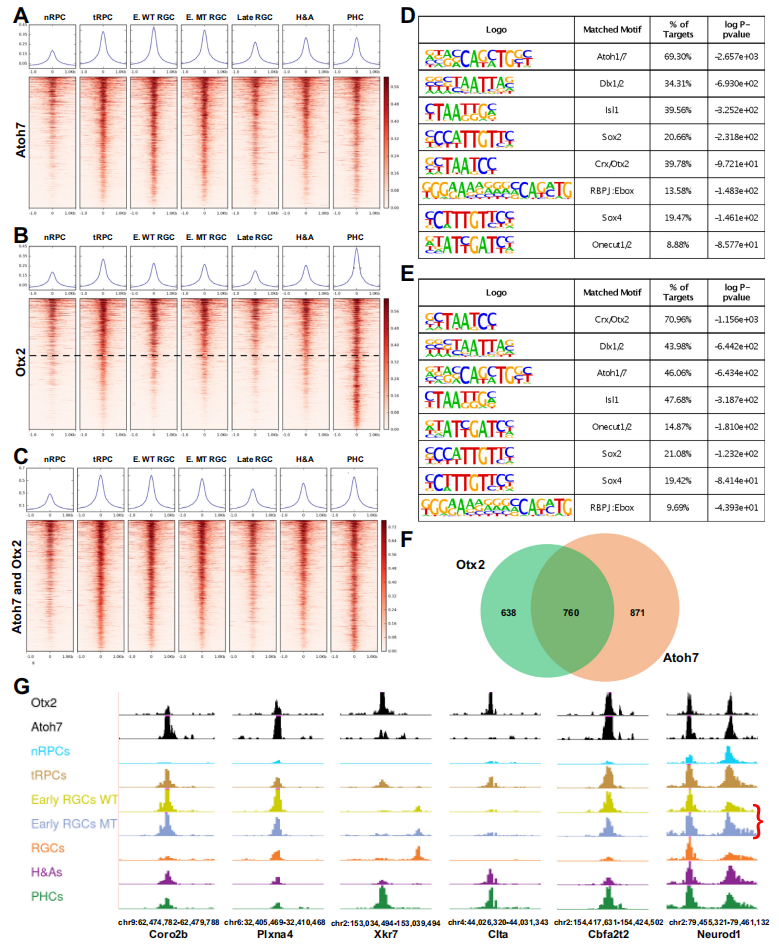
Fig4. DNA motif富集和足迹分析揭示了关键转录因子的细胞状态/类型特异性占用 5. 过渡性RPCs(tRPCs)的表观遗传景观 通过scRNA-seq发现,所有视网膜发育轨迹都经历了一个被称为神经发生性或过渡性RPCs(tRPCs)的共同状态。tRPCs的特征是表达编码Notch通路组分的基因,包括Dll1、Dll3、Dll4、Notch1、Hes5、Mfng和Rpbj,以及共表达编码不同谱系转录因子的基因。tRPCs具有一组独特的活性增强子,这些增强子可能支持该细胞状态中基因的表达(Fig2A)。与nRPCs相比,tRPCs的表观遗传状态中最显著的变化是bHLH转录因子和Otx2识别的DNA结合位点的显著富集(Fig4A,4B)。多个bHLH转录因子在tRPCs中表达,这些因子在视网膜细胞分化中发挥着不同的作用。E box结合位点在nRPCs中并未富集,但在早期RGCs、H&As和PHCs中持续富集。Otx2 DNA结合位点在tRPCs中也富集,并在PHCs中保持,但在其他分化的神经元中不保持。Atoh7和Otx2驱动tRPCs朝着两种不同的细胞命运发展,了解它们如何与tRPC表观遗传景观相互作用和影响可能有助于揭示这两个相应谱系的出现方式。通过CUT&Tag鉴定了Atoh7和Otx2在E14.5视网膜中结合的基因组位点,发现这些增强子可能介导这两个转录因子的作用。与Atoh7结合的增强子在tRPCs和早期RGCs中最活跃,而与Otx2结合的增强子在tRPCs和PHCs或仅PHCs中最活跃(Fig5A,5B,5G)。这些增强子的活性与这两个因子在视网膜细胞分化中的作用一致。通过HOMER结合位点富集分析,验证CUT&Tag确实鉴定出了这两个转录因子的结合位点(Fig5D,5E)。与Atoh7和Otx2相互作用的增强子的活性进一步确立了它们在tRPCs和相应发展中的神经元RGCs和PHCs中的功能。
Fig5. Atoh7和Otx2结合不同但重叠的增强子 6. Atoh7和otx2相互交叉调节下游基因,驱动tRPCs走向两个不同的谱系 在视网膜细胞分化过程中已知的Atoh7和Otx2这两个转录因子与其他转录因子合作,共同调控下游基因,包括在nRPCs中作用的Sox2,tRPCs中的SoxC因子和Onecut因子,以及分化神经元中的Dlx1/2和Isl1。此外,Atoh7和Otx2结合的增强子中富集了Rbpj和E box的复合结合位点,可能介导Notch通路在建立tRPC状态中的作用。Atoh7和Otx2的增强子集存在显著重叠,这些增强子在tRPCs、早期RGCs和PHCs中活跃(Fig5D,5E)。研究还发现Atoh7和Otx2共同调控了许多关键的调节基因(Fig5F,6,9),包括参与RGC和光感受器分化的基因。这些发现表明Atoh7和Otx2在细胞分化早期相互作用并共同调控靶基因,但随着tRPCs分化为不同细胞类型,它们的功能也有所分歧。Atoh7和Otx2在调控靶基因表达中的贡献可能取决于基因和细胞背景。它们可能在某些情况下共同激活靶基因,如Notch通路基因。Atoh7直接调控这些基因,而Otx2可能与Atoh7合作。在特定细胞谱系基因中,它们可能起着相反的作用,促进不同谱系的发展。Atoh7促进RGC基因表达,抑制PHC基因,而Otx2则相反。这种交叉调控可能是多能tRPCs产生不同细胞命运的一般机制。
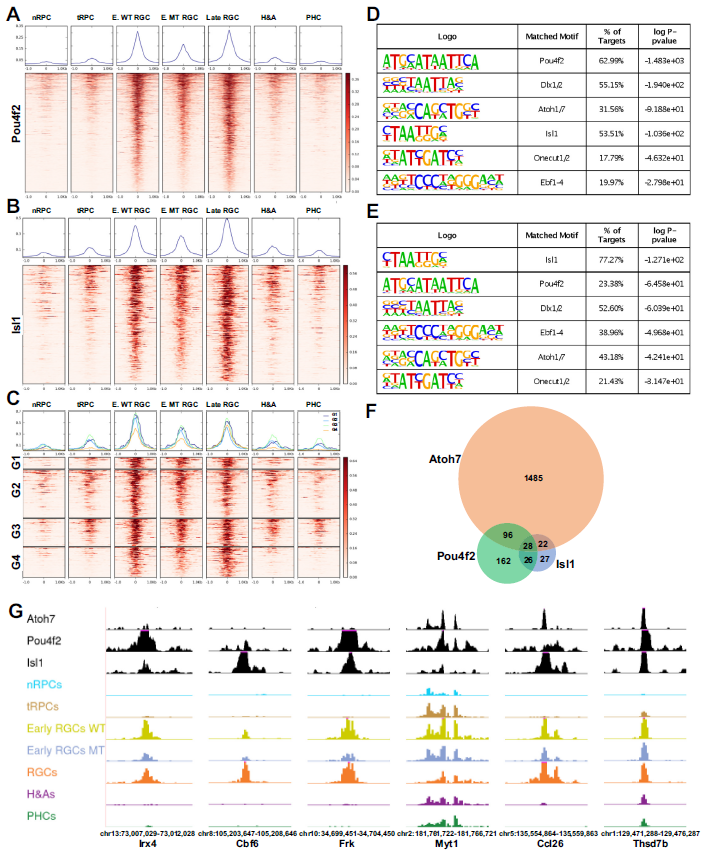
Fig6.由Atoh7和Otx2共同调控的示例基因 7. Atoh7, pou4f2和isl1与表观基因组相互作用,以顺序和组合的方式激活RGC特异性基因 视网膜神经节细胞(RGC)谱系的建立涉及到一系列基因调控级联反应,包括Atoh7在早期tRPCs和RGCs中的作用,以及Pou4f2、Isl1等转录因子在早期和晚期RGCs中的作用。研究发现Atoh7仅对特定一部分增强子的活性起调控作用,与其相关基因包括多个参与RGC谱系的关键调控基因。通过CUT&Tag实验,发现Pou4f2和Isl1分别与1535个和313个增强子结合,这些增强子在RGC谱系中高度特异且活性较高。Pou4f2和Isl1结合的增强子富含其他转录因子的结合位点,包括Dlx1/2、Atoh7、Ebf1-4和Onecut1/2。这些结果表明这些因子确实调节了Pou4f2和Isl1的功能(Fig7A,7B)。通过HOMER鉴定出的Pou4f2和Isl1结合增强子富集程度最高的motif与之前报道的motif一致,再次证实了CUT&Tag鉴定出了它们的真正结合位点(图7D, E)。在Atoh7缺失的早期RGCs中,Pou4f2和Isl1结合的增强子活性明显降低,但仍比其他细胞状态/类型活性高(Fig7A,B,C,G)。这些发现揭示了Atoh7、Pou4f2和Isl1等因子在调控RGC谱系形成中的作用机制,为进一步理解视网膜发育提供了重要线索。
Fig7. Pou4f2和Isl1结合RGC特异性增强子 研究发现,Atoh7、Pou4f2和Isl1这三个因子结合的增强子中包含许多已知与它们下游基因相关的基因,进一步验证了研究结果的有效性。此外,这三组相关基因有很大重叠(Fig.7F)。Atoh7、Pou4f2和Isl1在RGC轨迹的早期和晚期阶段发挥作用,它们对不同增强子的差异结合以及在RGC轨迹上的动态变化,揭示了这些转录因子通过顺序和组合作用激活单个RGC基因的不同调控方式(Fig.8)。研究还指出,Atoh7参与了许多早期和部分晚期RGC基因的初始激活,而Pou4f2和Isl1则在Atoh7关闭后维持早期RGC基因的表达,并激活许多晚期RGC基因的表达。另外,其他转录因子如Ebf1-4和Onecut1/2可能与Pou4f2和Isl1合作,共同调控RGC基因的表达。研究还发现,Atoh7和SoxC因子可能在促进RGC谱系从tRPCs中分化过程中相互激活,特别是Sox11与Atoh7和Pou4f2结合的增强子有关。这些发现揭示了Atoh7和SoxC因子可能在激活RGC分化程序中相互交叉激活。
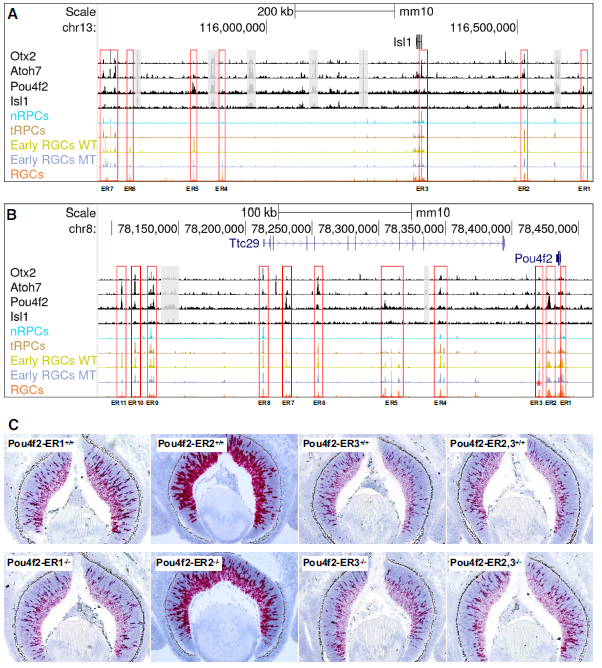
Fig8. Atoh7、Pou4f2和Isl1对RGC特异性基因的序列和组合调控 8.控制isl1和pou4f2表达的表观遗传机制 由于Isl1和Pou4f2是在RGC发育过程中最早激活的谱系特异性基因,并且它们在建立RGC谱系中的作用已经明确,因此破译这两个基因的激活机制对于理解RGC如何从多能tRPC状态出现至关重要。研究利用scATAC-seq和CUT&Tag数据详细研究了这两个基因可能的调控机制。研究发现,通过分析周围增强子的活性和Otx2、Atoh7、Pou4f2和Isl1的结合位点,确定了可能调控Isl1表达的七个主要增强子区域(ER1-7)(Fig.9A)。这些增强子的活性动态与结合的转录因子的功能密切相关。类似地,研究还鉴定了可能调控Pou4f2的十一个增强子区域(ER1-11),它们的活性动态与Pou4f2表达密切相关(Fig.9B)。Isl1和Pou4f2相关的增强子特征表明这两个基因通过Atoh7和Pou4f2/Isl1的类似顺序功能激活,Atoh7首先通过与Otx2竞争并结合到与这两个基因相关的特定增强子上,导致它们的初始激活。一旦表达,Pou4f2、Isl1和可能其他转录因子会结合到不同但有重叠的一组增强子上以维持它们的表达。对候选增强子对Pou4f2和Isl1表达的贡献可以进行实验来检测。研究者通过CRISPR/cas9删除了三个Pou4f2增强子区域(ER1-3),并通过RNA-scope原位杂交检测了E12.5, E14.5和E17.5对Pou4f2表达的影响(Fig.9B, C)。结果显示ER1和ER2对Pou4f2表达的维持起着重要作用,而ER3可能起着抑制作用。这些结果表明Pou4f2在RGCs中的空间和时间表达是由多个增强子的综合和可能冗余的输入决定的。
Fig9. 早期RGC特异性表达Isl1和Pou4f2的调控机制 本文利用scATAC-seq和CUT&Tag技术,研究了视网膜细胞分化过程中的表观遗传景观变化,发现不同细胞状态的增强子活性差异可将视网膜细胞分组,并揭示了细胞发育轨迹中基因表达与表观遗传景观的关系。从nRPCs到tRPCs再到特定神经元的发育轨迹中,细胞状态特异性增强子的活性变化导致基因表达变化。转录因子在调控表观遗传景观和基因表达中发挥关键作用。研究结果支持关键转录因子在推动细胞向不同谱系发展中既合作又竞争的模式。CUT&Tag与scATAC-seq数据的交集揭示了增强子动态及其关联基因,为进一步探究转录因子与表观基因组互作机制奠定基础。
Fig10.从过渡时期的RPCs中出现RGC和PHC谱系的模型 探索细胞内的奥秘,解锁基因表达的密码! 爱基百客拥有10年深耕表观遗传学研究服务的经验,我们的表观组学和单细胞测序平台技术已十分完善。我们提供从大规模(Bulk级别)的CUT&Tag、ATAC-seq、ChIP-seq到单细胞级别的scATAC-seq、scChIP-seq等一系列高精度的表观遗传学测序服务。我们的服务范围涵盖实验设计、样本处理、数据分析到验证的全流程,旨在为客户提供全面、专业的技术支持。如果您在表观遗传学研究领域有任何技术需求,欢迎随时联系我们,我们期待为您的科研工作助力!
项目咨询 了 解 更 多 { 往 期 精 彩 回 顾 } 精选合集,欢迎收藏哟! |