NEWS
新闻资讯
|
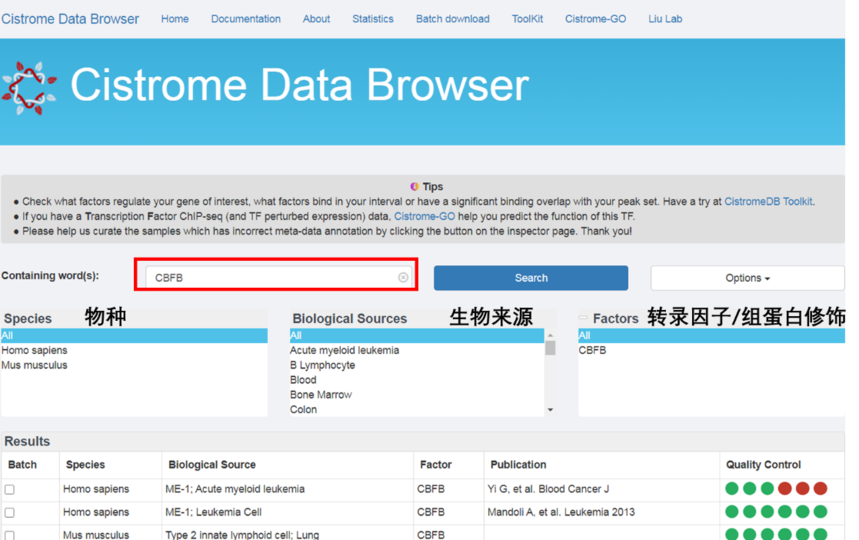
转录因子/组蛋白修饰靶基因数据库:Cistrome DB使用教程最近有小伙伴经常询问怎么预测转录因子的下游靶基因,以及预测一些组蛋白修饰影响的靶基因信息。今天就给大家介绍一下Cistrome数据浏览器(Cistrome Data Browser)。 Cistrome DB是来自人类和小鼠的ChIP-seq、ATAC-seq和DNase-seq数据的资源,提供了转录因子、辅因子、染色质重塑因子、组蛋白翻译后修饰和可用于核酸内切酶活性的染色质区域的全基因组位置图。Cistrome DB包含大约45000个人类和44000个小鼠样本;我们可以通过关键字、菜单和数据驱动的搜索工具找到信息丰富的染色质图谱。 浏览器搜索功能可以预测查询基因的调节因子以及潜在顺式调节元件的细胞类型和因子依赖性功能。而且网站还有toolKit可以检索哪个因子调控了我们感兴趣的基因、哪些因子结合在感兴趣的区域;查看哪些转录因子的结果与输入peak的结果有明显的重叠,用于转录因子的colocation分析。Cistrome-GO还可以做ChIP数据集靶基因的功能富集分析。 话不多说,我们来开始操作吧。 网站:http://cistrome.org/db/#/ 打开界面。我们可以在红框里输入感兴趣的转录因子,以CBFB为例。 Species代表物种,Biological Sources 代表生物来源,Factors代表转录因子/组蛋白修饰类型。
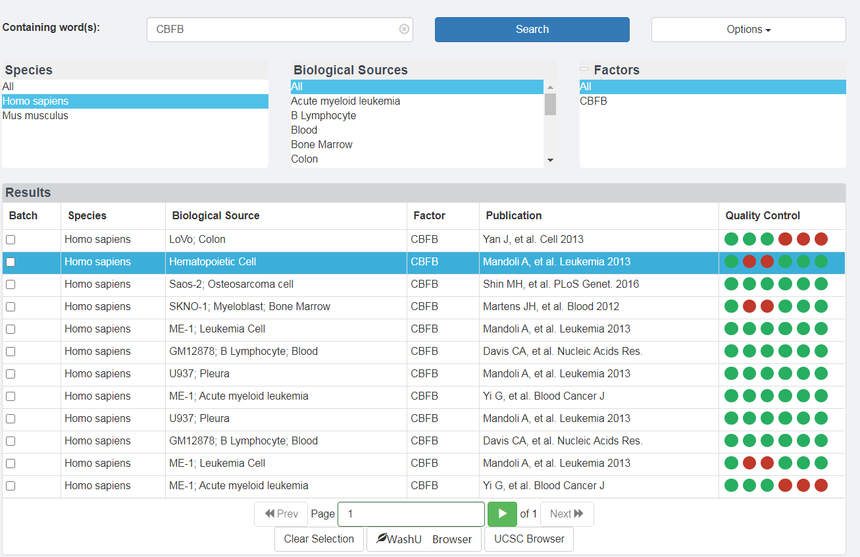
这里选择人的造血细胞CBFB的结果进行展示:
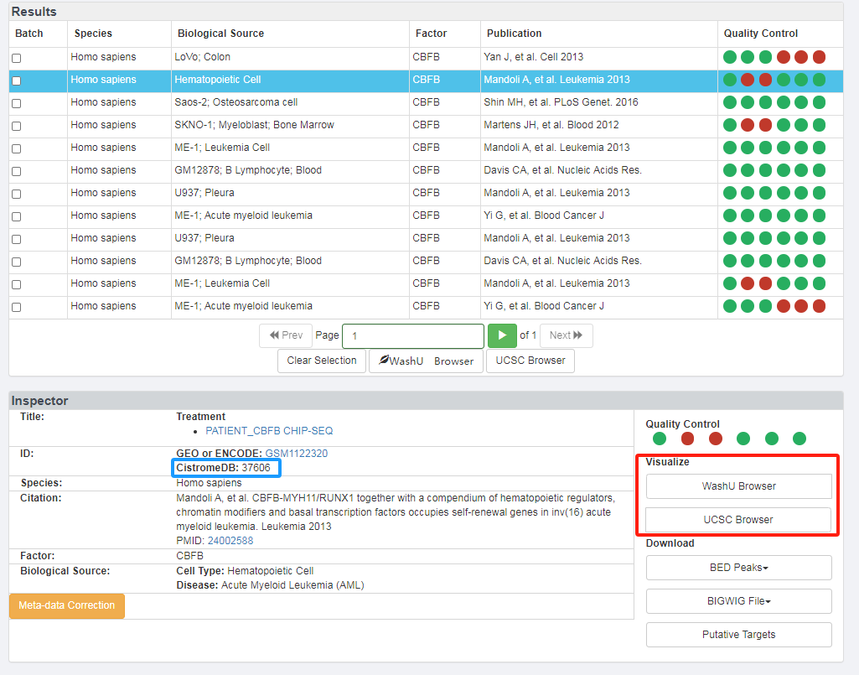
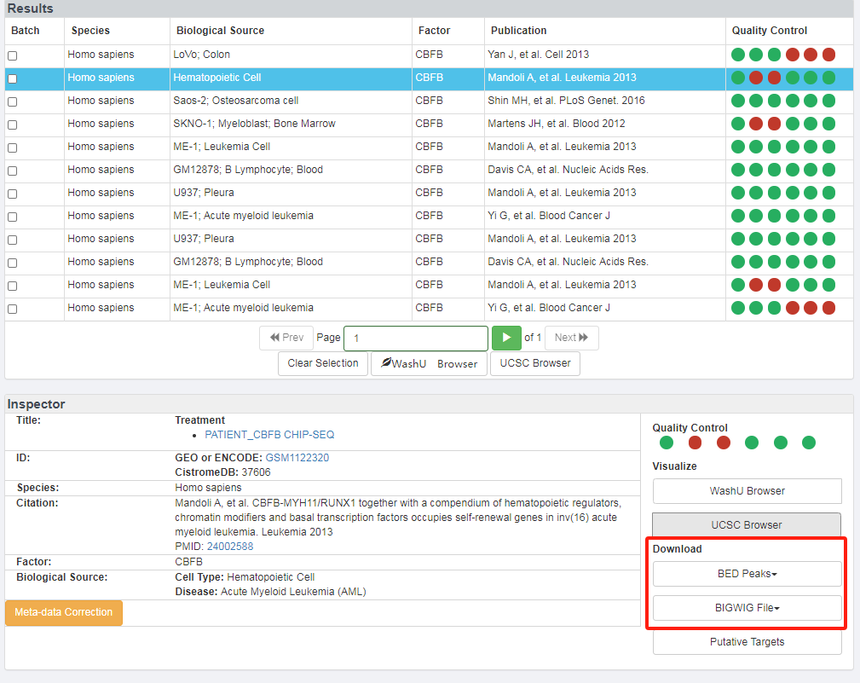
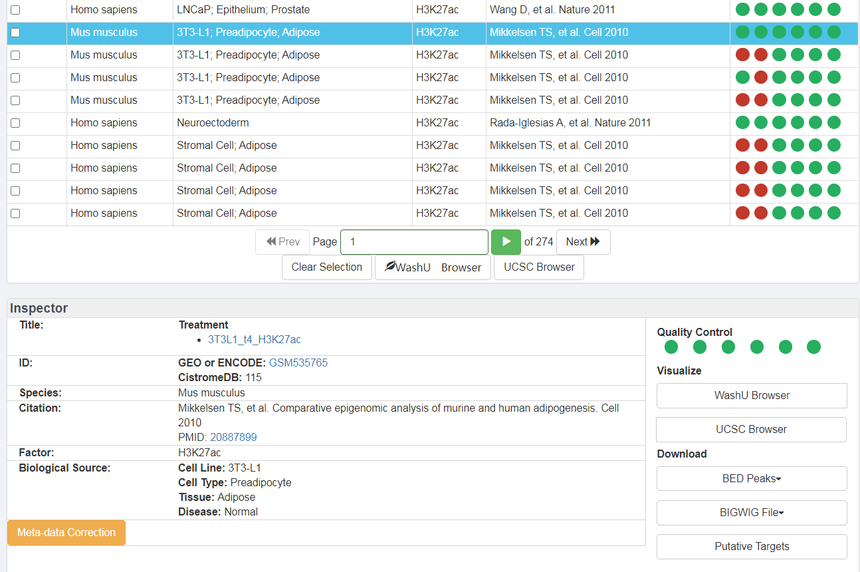
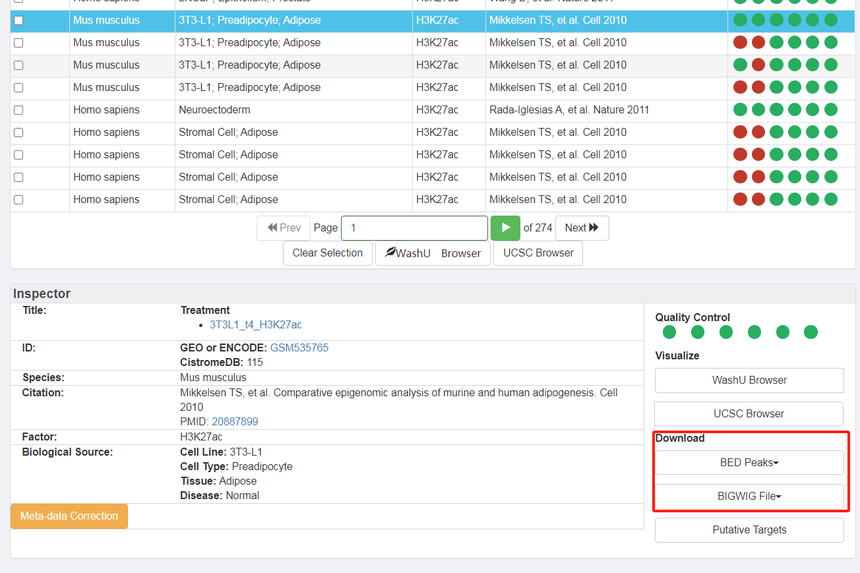
每个ChIP-seq和DNase-seq样本都有一个唯一的数据集ID, Cistrome DB为每个数据集(包含手动管理的元数据)进行注释,包括物种、因素、生物来源、发布时间和处理状态。点击单个数据集可获取分析结果和质控指标,点击多个数据集可提取批量数据查看,选择感兴趣的数据集后我们可以将数据发送到genome browser(WashU Browse:、UCSC Browse)进行联合分析,如辅因子、染色质调节因子与组蛋白修饰之间的关系等。 Result下面还有Inspector部分,在红框地方,我们可以点击WashU Brower以及UCSC Browser进行浏览。需要注意的是篮框,我们用到的是CistromeDB:37606的数据。
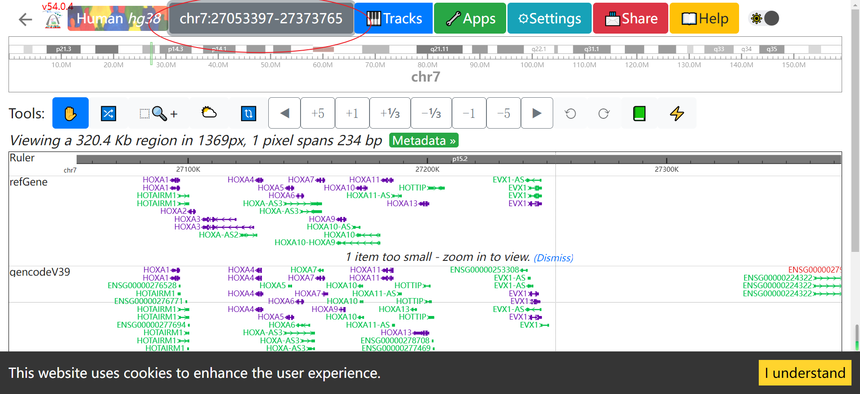
1. 点击WashU Browser显示如下:
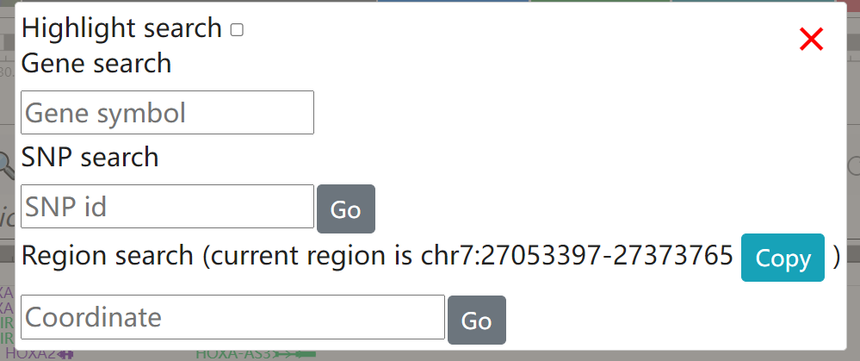
我们可以点击黑灰色部分(上图红色圈圈),弹出如下窗口,进去可以搜索感兴趣的靶基因。
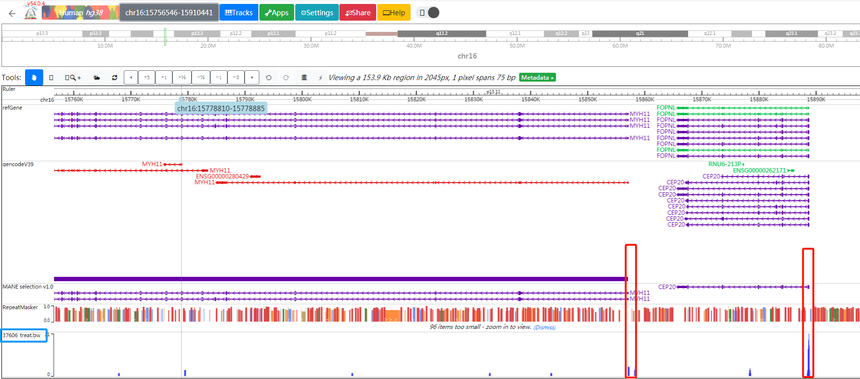
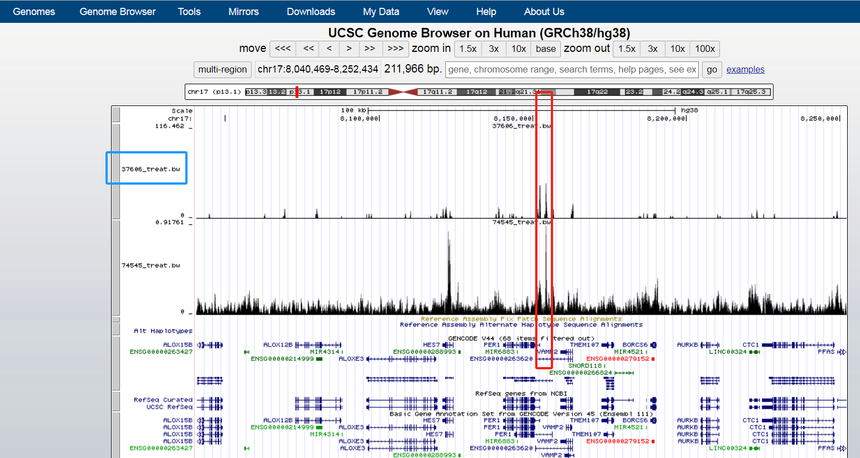
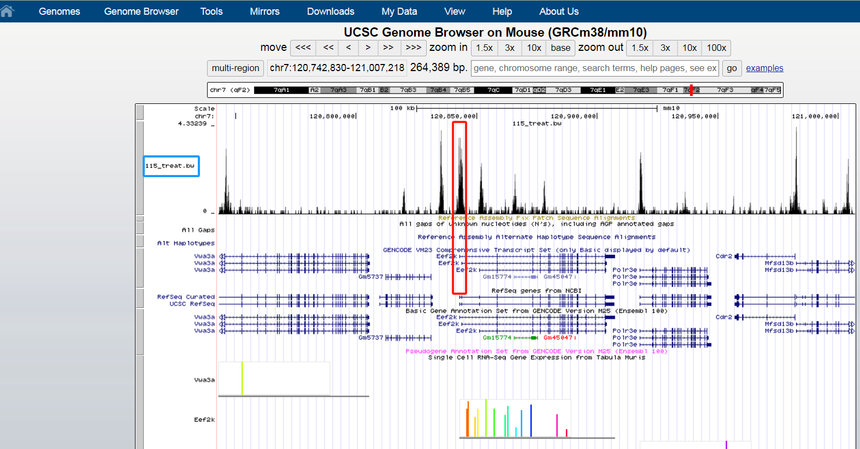
我们也可以直接选中界面区域,进行左右拖动,看整个基因组的可视化情况。下图界面展示CEP20和MYH11的基因可视化情况,可以看到这两个基因的启动子区域都有peak(红框展示)。其中基因上的箭头表示基因的方向性。
2. 点击UCSC Browser 直接可以搜索CEP20或MYH11,得到同样的可视化情况。也可以通过点击move的<>等箭头来看整个基因组上的可视化情况。
3. 还可以点击Downlod的BED Peaks 以及BIGWIG File部分进行下载,后用IGV可视化进行浏览(公众号往期文章详细介绍了IGV使用教程)。
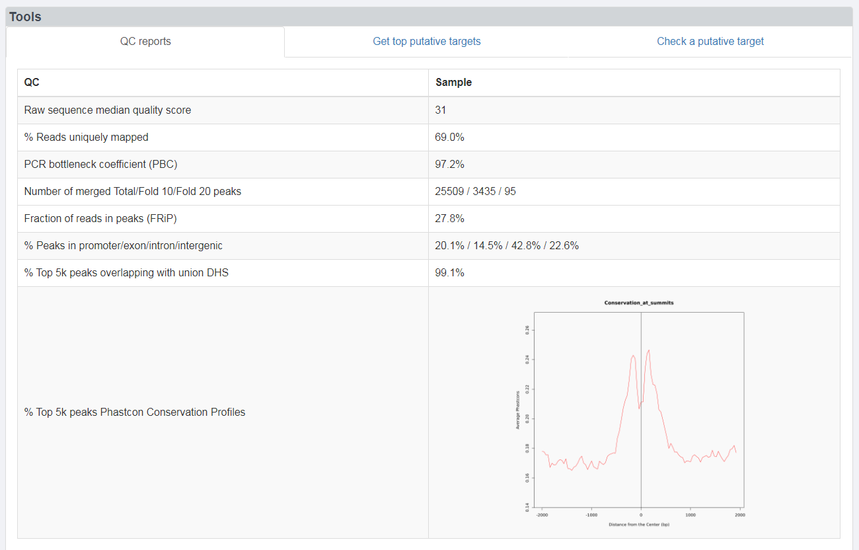
找到感兴趣的靶基因后,根据peak的位置提取peak序列后就可以设计引物进行验证了。不过这也只是其他研究者对应的细胞材料的数据,建议大家自己研究还是从ChIP-seq开始,再做ChIP-qPCR,这样更好一些哦。 另外Tools部分还展示了QC reports,QC motifs,Get top putative targets,Check a putative target。 QC reports可以查看数据质量情况:
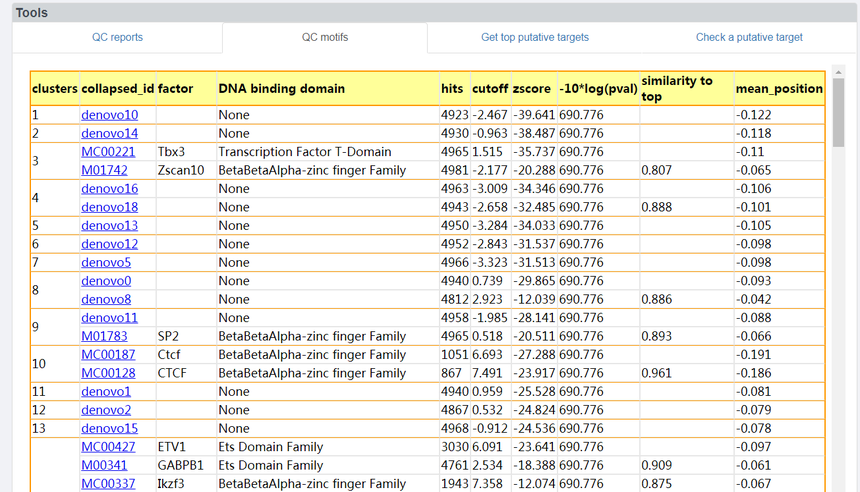
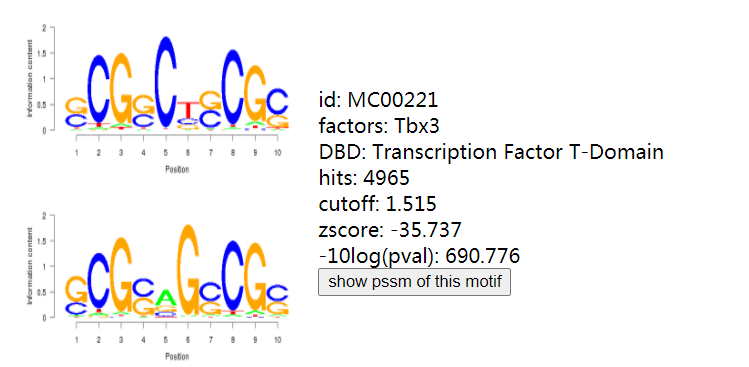
Motif信息
可以点击查看详细的motif
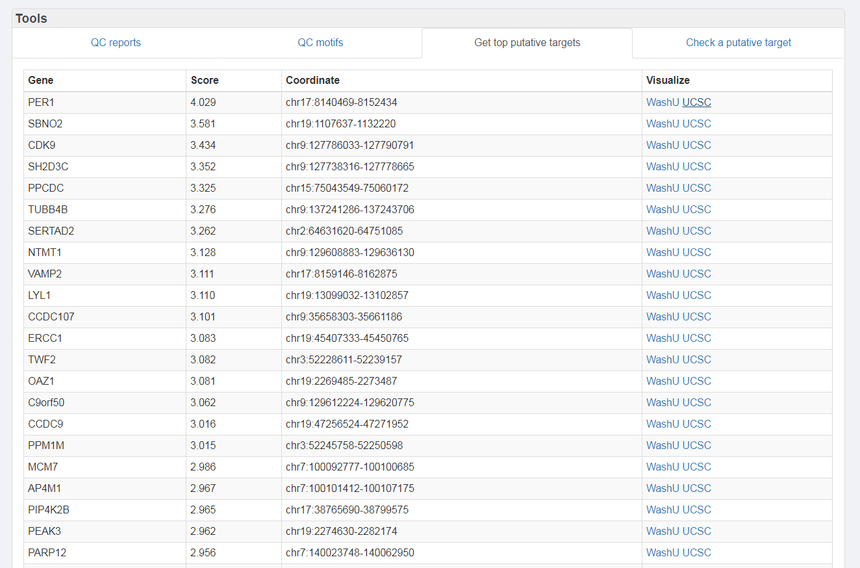
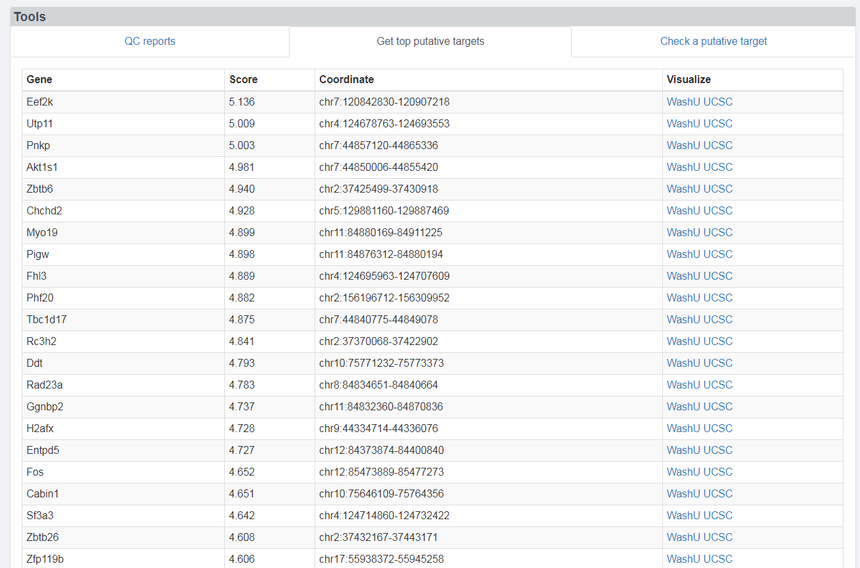
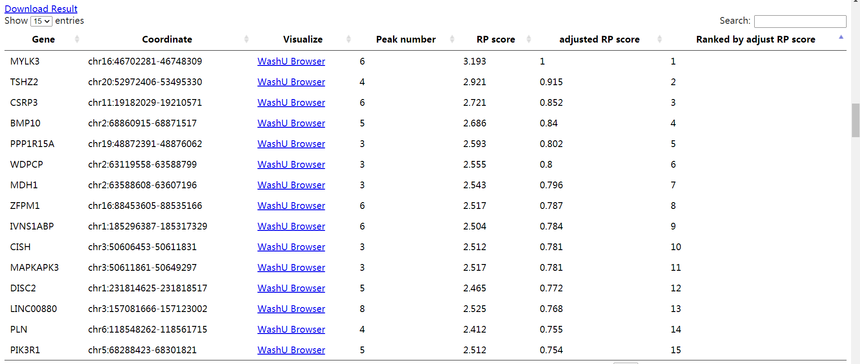
Get top putative targets 得到排分靠前的靶基因,可以用WashU以及UCSC浏览
点击PER1对应的可视化浏览器得到以下结果:
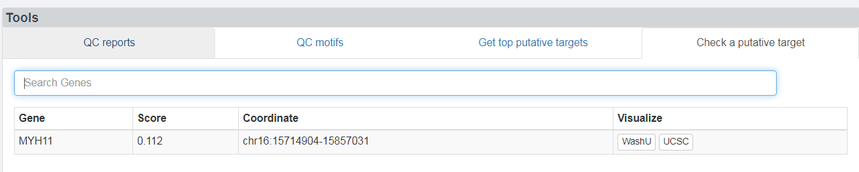
Check a putative target 查找感兴趣的靶基因,搜索MYH11得到该基因的得分、位置等信息。
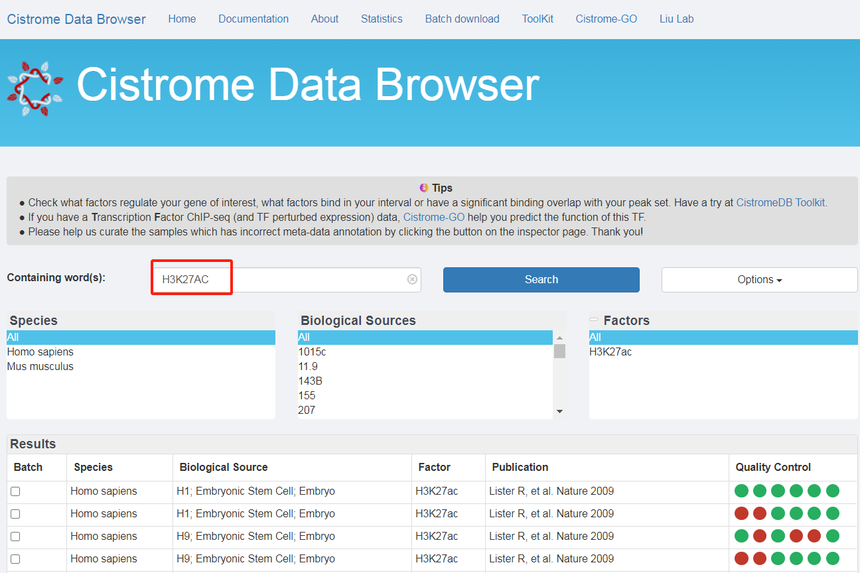
在红框里输入H3K27ac
以3T3-L1(用于贮存脂肪的前脂肪细胞)为例; 点击后
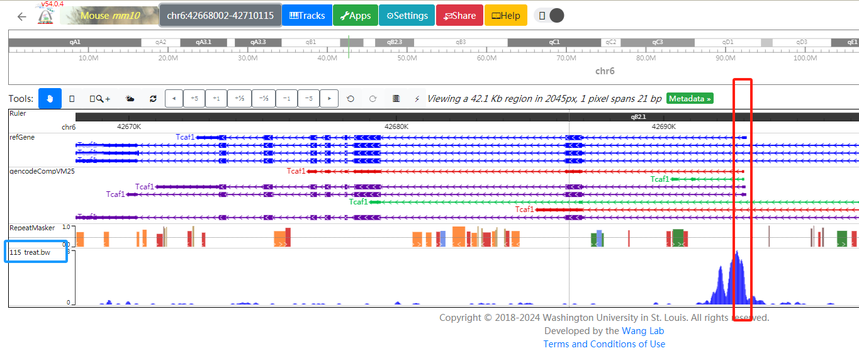
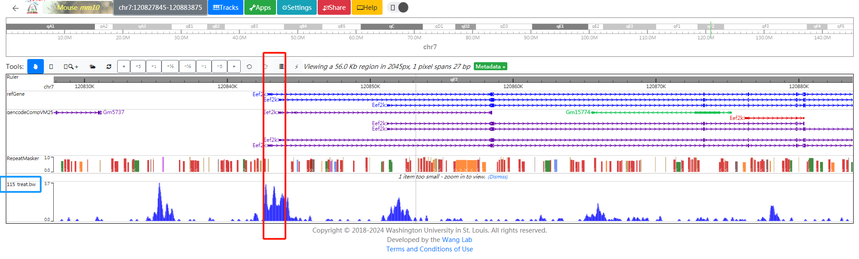
1. 点击WashU Browser显示: 搜感兴趣的基因,以Tcaf1为例;可以看到在启动子区域有明显的peak。
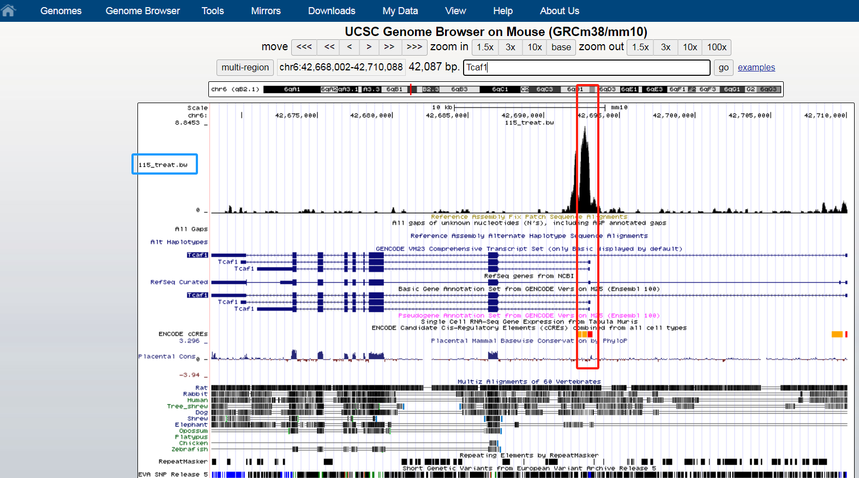
2. 点击UCSC Browser 也定位到Tcaf1这个基因,可以看到同样的peak
3. 还可以点击Downlod的BED Peaks 以及BIGWIG File部分进行下载,后用IGV可视化进行浏览。
另外Tools部分: QC reports可以查看数据质量情况:
Get top putative targets 得到排分靠前的靶基因,可以用WashU以及UCSC浏览
点击Eef2k,看对应的可视化,结果如下:
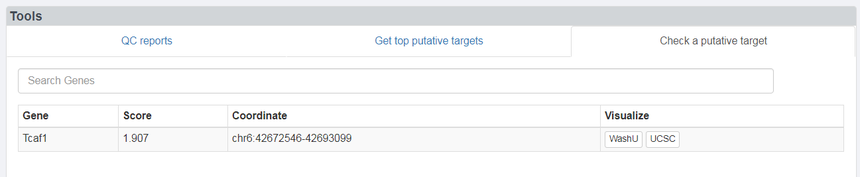
Check a putative target 查找感兴趣的靶基因;搜索得到Tcaf1该基因的得分、位置等信息
点击主页面的ToolKit
可以进入以下界面:
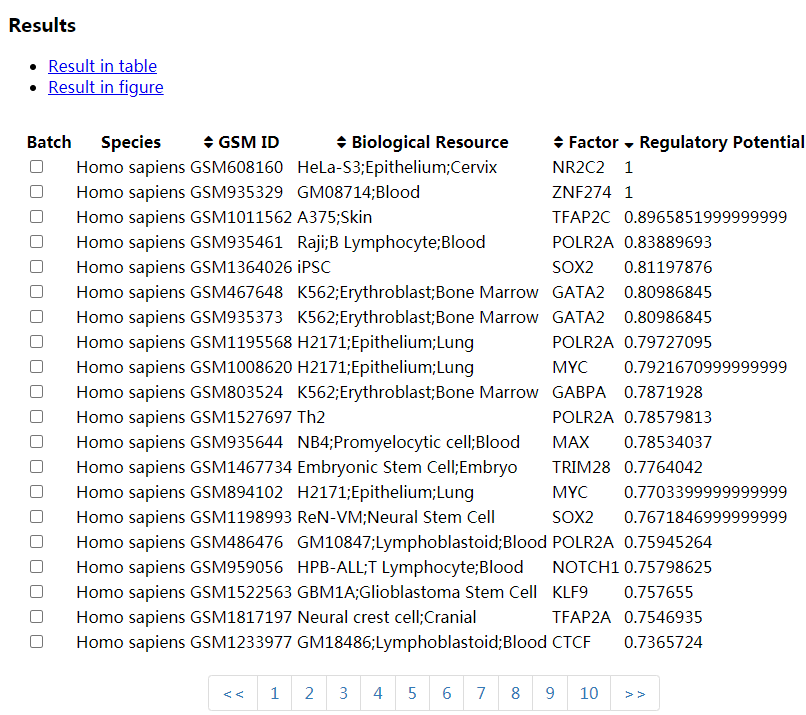
在第一个找哪些转录因子靶向感兴趣的基因中,输入E2F1为示例,得到结果如下: 可以看到有很多预测到的转录因子靶向E2F1。
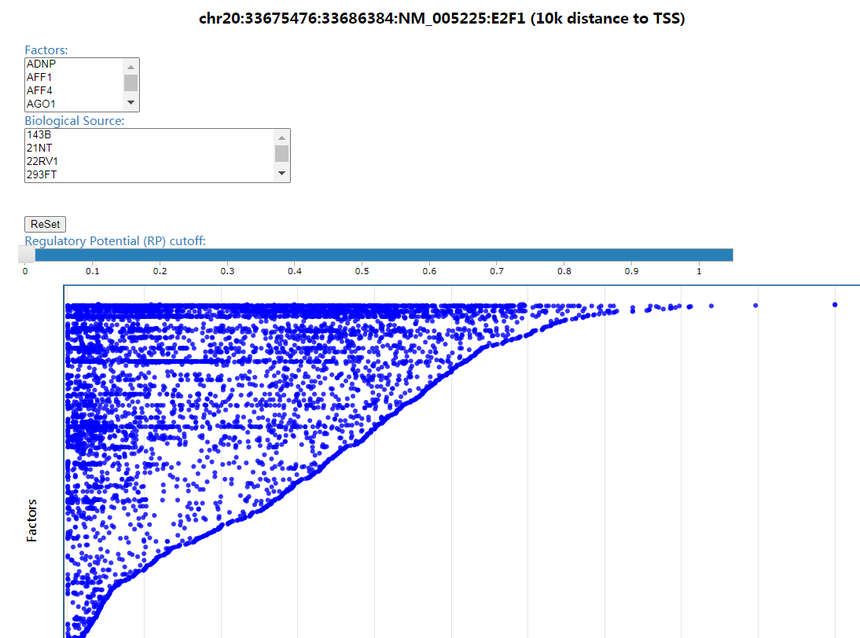
点击Result in figure得到以下信息:
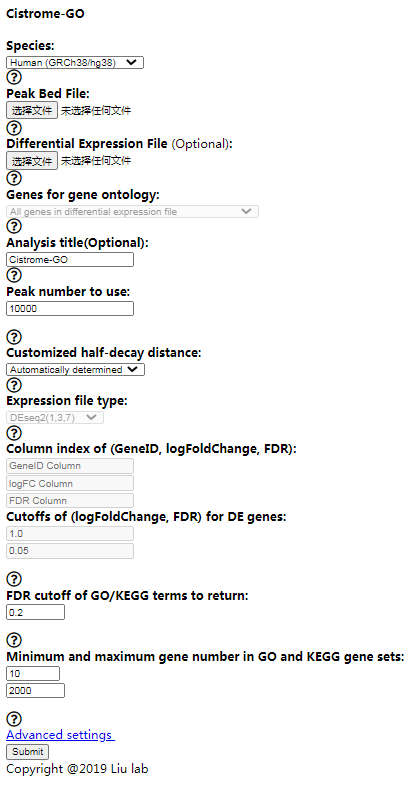
为了识别TF直接调控的基因,该网站还开发了一个名为Cistrome GO的网络服务器,该服务器允许用户输入ChIP-seq峰值,并通过加权基因周围峰值的贡献来输出基因得分,以指示TF的调控潜力。重要的是,Cistrome GO使用计算的全基因组评分进一步进行基因本体分析和途径富集。 它有两种工作模式。如果用户同时提供了TF的ChIP-seq文件和差异表达分析文件(基于TF),则Cistrome-GO将基于两种数据类型的整合执行集成模式分析。如果我们仅上传TF ChIP-seq文件,则Cistrome-GO将以单独模式执行分析。 我们可以导入peak的bed文件进行分析。
这里我们直接看一下示例的demo吧
选择GATA4 ChIP的数据
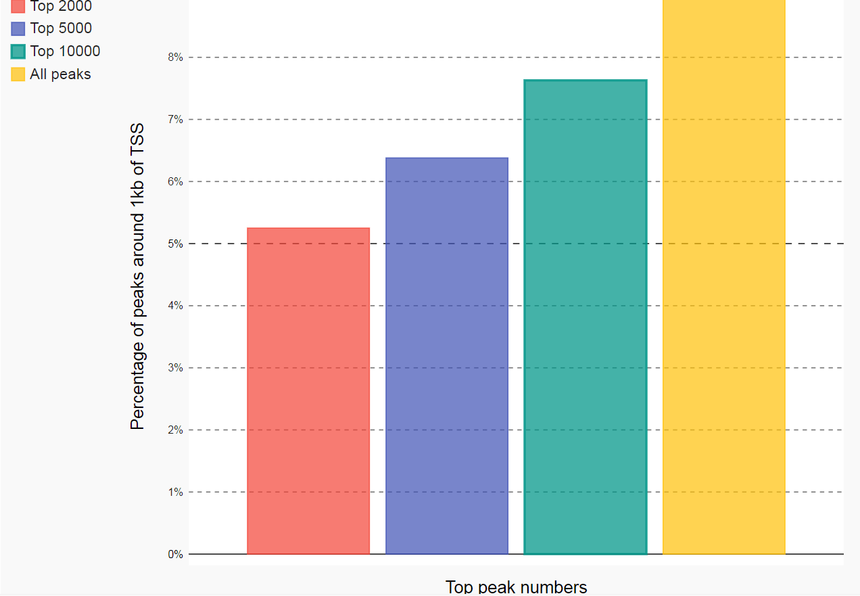
显示的是72934_peaks analysis 前10000个peak
8.941%的峰位于启动子区。这小于20%启动子类型阈值,因此衰减距离设置为10.0kb,适合于增强子类型分析。衰减距离可以在选项中指定。
显示关联基因的信息:
KEGG富集结果
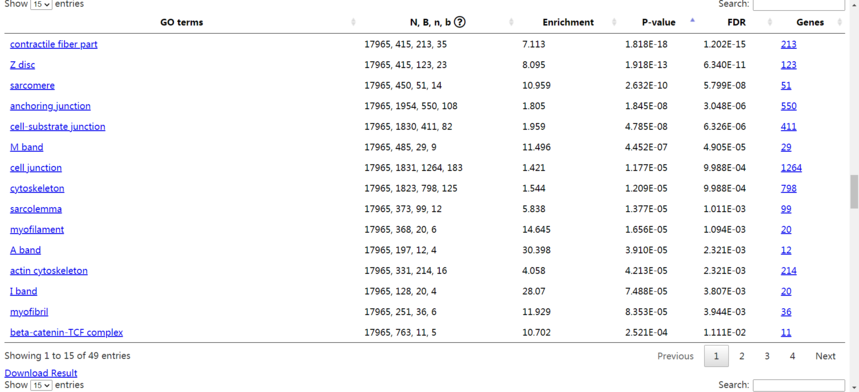
GO结果
总之,Cistrome DB可以找我们感兴趣的转录因子以及组蛋白修饰位点的靶向基因信息,还可以通过toolKit进行可以检索哪个因子调控了我们感兴趣的基因、结合在该区域;查看哪些转录因子的结果与输入peak的结果有明显的重叠,可以用于转录因子的colocation分析。Cistrome-GO还可以做ChIP数据集靶基因的功能富集分析。大家是不是心动了,赶紧用起来吧,顺便提一下做ChIP、CUT&Tag找爱基百客哦,目前CUT&Tag还在春季大促哦。
项目咨询 了 解 更 多 { 往 期 精 彩 回 顾 } 精选合集,欢迎收藏哟! |