NEWS
新闻资讯
|
干货 | 单基因甲基化:五大技术比拼,你pick哪个?
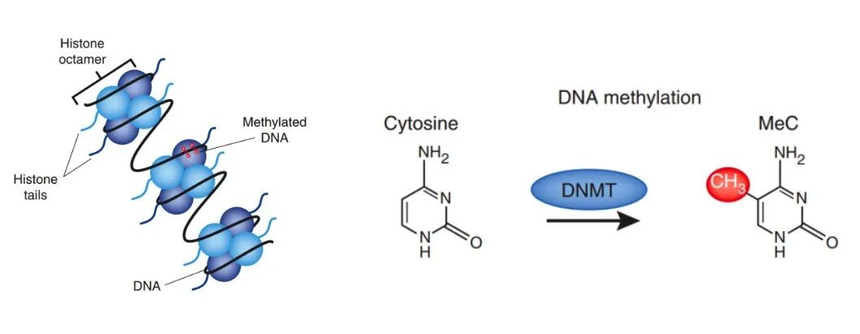
DNA甲基化是一种重要的表观遗传修饰方式,它在基因表达调控、细胞分化、发育和疾病发生等过程中发挥着重要作用。一般研究中所涉及的DNA甲基化主要是指发生在CpG位点(胞嘧啶-磷酸-鸟嘌呤位点,即DNA序列中胞嘧啶后紧连鸟嘌呤的位点)中胞嘧啶上第5位碳原子的甲基化过程,其产物称为5-甲基胞嘧啶(5-mC)。5-mC广泛存在于植物、动物等真核生物基因组中,是目前研究最多的一种DNA甲基化修饰形式。
这种化学修饰在多种生物过程中扮演着关键角色,包括胚胎发育、X染色体失活、基因沉默以及肿瘤发生等。因此,对DNA甲基化的研究不仅有助于我们理解生命现象的复杂性,也为疾病的诊断和治疗提供了新的视角。
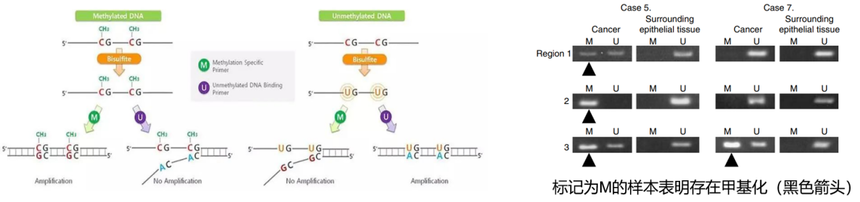
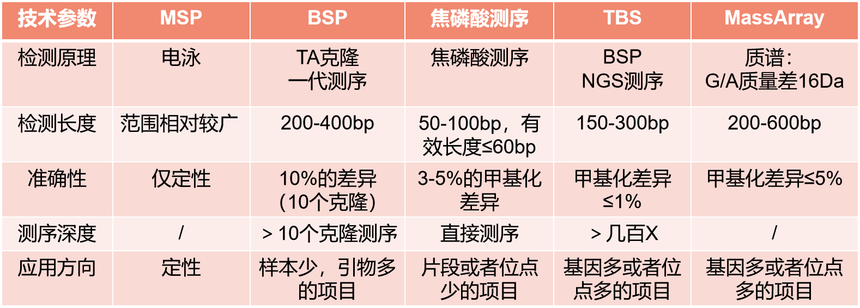
DNA甲基化对生命过程的影响 在 DNA 甲基化的研究领域中,多种技术和方法已被报道和应用。其中,全基因组甲基化测序技术,如 WGBS、RRBS、meDIP 等,虽然具有高度的准确性和全面性,但其成本相对较高,限制了其在某些小规模研究或特定基因甲基化检测中的应用。因此,单基因甲基化以其相对较低的成本和灵活性,成为了一种重要的甲基化研究工具(也称靶向甲基化检测技术)。下面带大家来看一下常见的单基因甲基化检测技术。 1. 甲基化特异性PCR——MSP MSP(Methylation-specific PCR)是一种简便快捷的甲基化检测技术,用于单基因甲基化分析。其基本原理是在对DNA进行亚硫酸氢盐处理后,针对处理后的DNA片段设计两对特异性引物。其中一对引物(引物对M)能与处理后的甲基化DNA链结合,而另一对引物(引物对U)则与处理后的非甲基化DNA链结合。如果第一对引物能够成功扩增出DNA片段,则表明该检测位点存在甲基化;若第二对引物能扩增出片段,则表明该位点不存在甲基化。MSP技术无需特殊仪器,通过电泳即可进行检测。然而,MSP技术仅能提供甲基化的定性信息,无法进行精确的定量分析。
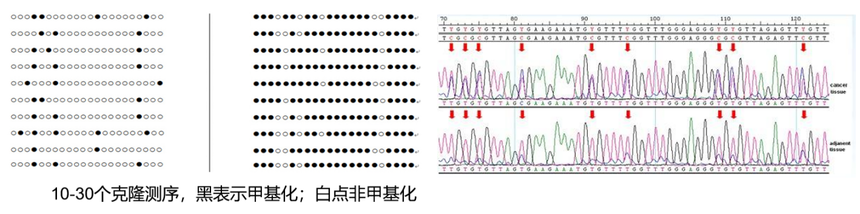
MSP技术原理和结果 2. 亚硫酸氢盐测序 PCR——BSP BSP(Bisulfite Sequencing PCR)是检测基因甲基化的经典方法,也是基于第一代测序技术对Bisulfite 处理后DNA 进行测序从而检测甲基化的技术。BSP 是针对目的片段设计特异性引物后进行PCR,对BSP产物进行TA克隆,选择10-30个阳性克隆进行测序,计算甲基化水平。该技术的主要优势体现在其单碱基分辨率上,不仅能进行定性分析,还能实现定量分析。然而,其实验步骤繁多,成本高昂,且通量较小。
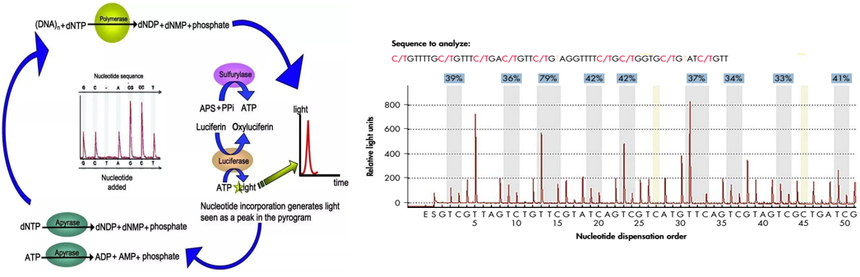
BSP技术原理和结果 3. 甲基化焦磷酸测序——Pyrosequencing 焦磷酸测序技术(Pyrosequencing)是由4种酶催化的同一反应体系中的酶级联化学发光反应。其原理是:引物与模板DNA退火后,在DNA聚合酶(DNA polymerase)、ATP硫酸化酶(ATP sulfurytase)、荧光素酶(1uciferase)和三磷酸腺苷双磷酸酶(Apyrase)4种酶的协同作用下,将引物上每一个dNTP的聚合与一次荧光信号的释放偶联起来,通过检测荧光的释放和强度,达到实时测定DNA序列的目的。该技术的主要优势在于灵敏度高,适合多种样本类型,但是其有效读长短,成本比较高。
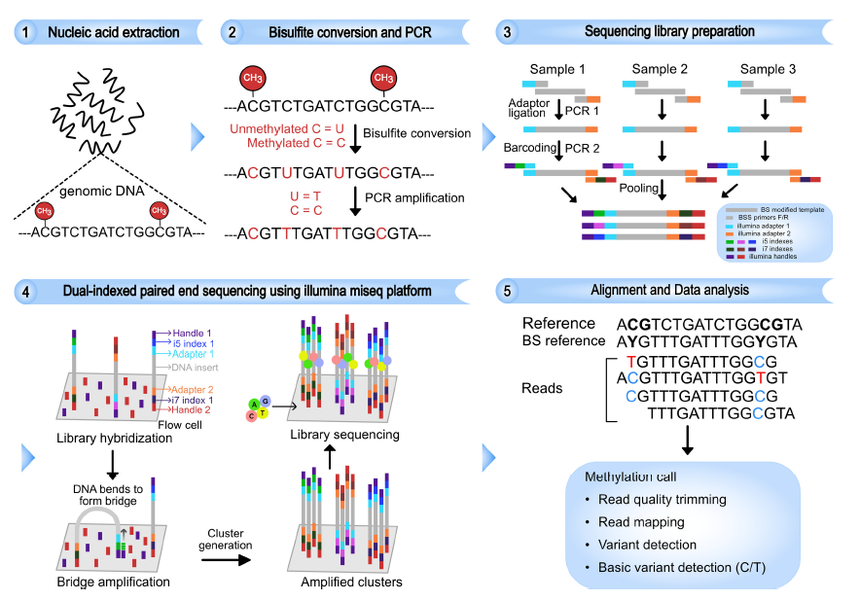
焦磷酸测序原理和结果 4. 高通量BSP测序——TBS TBS(Target Bisulfite Sequencing)是BSP结合NGS高通量测序验证基因甲基化的技术手段,对样本进Bisulfite 处理后,未发生甲基化的C变成U(PCR扩增后变为T),而甲基化的C保持不变,通过高保真耐U碱基的DNA聚合酶对Bisulfite处理的模版进行BSP 扩增,对来自同一样本的BSP扩增产物进行混合,并进行标签引物扩增,带上illumina 测序接头,最终获得每个样本带不同标签的测序文库,上机测序。经过优化设计的靶向Bisulfite测序技术,结合单碱基分辨率的高通量测序方法(覆盖度超过200倍),实现了高效、经济的大规模基因组甲基化状态分析,同时确保了检测位点的灵活性与准确性。
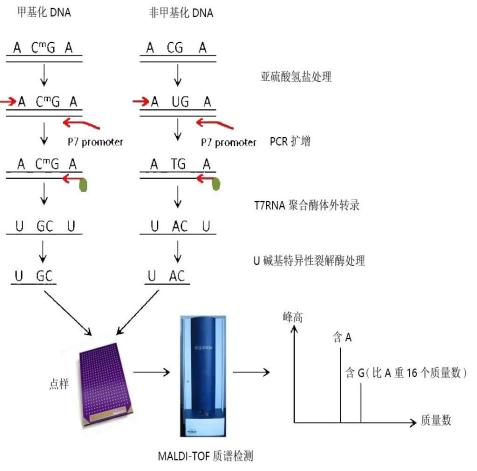
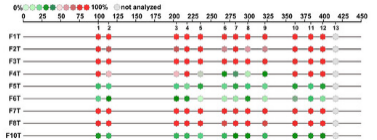
TBS原理和流程 目标区域甲基化大队列研究利器—TBS(往期推文详细介绍TBS)5. 飞行质谱法(MassArray) MassArray 技术通过碱基特异性酶切反应与灵敏可靠的MALDI-TOF质谱技术相结合,实现多重CpG位点分析。待测DNA经过Bisulfite处理,DNA中未甲基化胞嘧啶(C)转变为尿嘧啶(U),而甲基化修饰的胞嘧啶不变。利用5'末端带有T7-启动子的引物进行PCR扩增,产物经SAP虾碱性磷酸酶处理后用于碱基特异性的酶切反应。酶切后DNA片段的大小和分子量取决于亚硫酸盐处理后的碱基变化,质谱测定每个片段的分子量,软件EpiTYPER则能自动报告每个相应片段的甲基化程度。该技术适用1-20个片段的中通量验证,样本数量不受限;覆盖约400-600bp长度的区域位点,属于最长的locus-specific assays,准确性高,数据简便。
MassArray原理和结果 DNA甲基化 | MassArray质谱检测助力甲基化研究 (往期推文详细介绍MassArray)
单基因甲基化检测技术在生物学研究中具有广泛的应用前景。爱基百客作为国内领先的表观服务商,配备了完善的表观组学实验平台和高通量测序平台,能提供全基因组范围内的甲基化测序技术(WGBS、RRBS、meDIP等),也能提供单基因甲基化检测技术(TBS和MassArray)。我们可以提供从实验设计-样本处理-数据分析一站式服务,为您的科研工作提供全方位支持。如果您在表观遗传学研究领域有任何技术需求,欢迎随时联系我们,我们期待为您的科研工作助力!
项目咨询
1. Wan Q L, Meng X, Dai W, et al. N6-methyldeoxyadenine and histone methylation mediate transgenerational survival advantages induced by hormetic heat stress[J]. Science advances, 2021, 7(1): eabc3026. 2. Ding X, Liu X, Jiang G, et al. SlJMJ7 orchestrates tomato fruit ripening via crosstalk between H3K4me3 and DML2‐mediated DNA demethylation[J]. New Phytologist, 2022, 233(3): 1202-1219. 3. Chen M, Ruan R, Zhong X, et al. Comprehensive analysis of genome‐wide DNA methylation and transcriptomics between ovary and testis in Monopterus albus[J]. Aquaculture Research, 2021, 52(11): 5829-5839. 4. Huang C, Ying H, Yang X, et al. The Cardamine enshiensis genome reveals whole genome duplication and insight into selenium hyperaccumulation and tolerance[J]. Cell discovery, 2021, 7(1): 62. 5. Tan H, Tong X, Gao Z, et al. The hMeDIP-Seq identified INPP4A as a novel biomarker for eosinophilic chronic rhinosinusitis with nasal polyps[J]. Epigenomics, 2022, 14(12): 757-775. 6. Xu Q, Huang S, Guo G, et al. Inferring regulatory element landscapes and gene regulatory networks from integrated analysis in eight hulless barley varieties under abiotic stress[J]. BMC genomics, 2022, 23(1): 843. 7. Jiabu D, Yu M, Xu Q, et al. Genome-Wide DNA Methylation Dynamics During Drought Responsiveness in Tibetan Hulless Barley[J]. Journal of Plant Growth Regulation, 2023, 42(7): 4391-4401. 8. Shi Y, Yu B, Cheng S, et al. The Change in Whole-Genome Methylation and Transcriptome Profile under Autophagy Defect and Nitrogen Starvation[J]. International Journal of Molecular Sciences, 2023, 24(18): 14047. 9. Zou J J, Cai X, Yang J, et al. DNA hypomethylation mediates flower opening and senescence in sweet osmanthus through auxin and ethylene responsive pathways[J]. Postharvest Biology and Technology, 2023, 198: 112250. 了 解 更 多 { 往 期 精 彩 回 顾 } 精选合集,欢迎收藏哟! |